- Foro
- Foros sobre Micología de fungipedia
- Microscopía
- Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
 Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
- JOSEP
-
 Autor del tema
Autor del tema
- Fuera de línea
- Moderador
-

Menos
Más
- Mensajes: 8430
- Gracias recibidas: 8310
2 años 1 mes antes #108662
por JOSEP
Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm. Publicado por JOSEP
Hola a tod@s.
El único hongo que tenía pendiente de identificar de mi recogida de muestras conjunta con Juan Andrés, Javi y Aitor, del pasado 30 de Octubre por tierras Burgalesas.
En el momento que saqué la microscopía no conseguí llegar a ninguna propuesta convincente y no tengo perdón, ya que si bien la especie no la había encontrado anteriormente, si que ya la había estudiado de unas muestras que me había traído un amigo allá por 2018 y además ya la había identificado correctamente en su momento, en esta ocasión para salir de dudas decidí recurrir al análisis molecular, la secuenciación ITS determinó que se trataba con una coincidencia del 99.58% de Mallocybe malençonii OQ133558. La Mallocybe malenconii = Inocybe malenconii ya creo haberla estudiado y en mi trabajo las esporas presentaban un cociente Q de (1.9) 2 - 2.4 (2.6), con una media de Qe = 2.2 y en esta ocasión el cociente Q estaba en Q = (1.4) 1.5 - 1.7 (1.8), con una media de Qe = 1.6. Según todas las Claves se indica un cociente Q para la Mallocybe malenconii superior a 2, con lo cuál era del todo imposible que en esta ocasión se tratara de este.
A nivel molecular se considera que dentro de la misma especie puede haber una diferencia de hasta un 0,5%, y aunque la coincidencia del 99.58% se encontraba dentro de este rango, queda pero que muy claro que hay en Genbank muchas secuencias mal identificadas, tal y como me ha tocado en esta ocasión.
Al disponer de tiempo y reabrir el tema pronto he dado con la solución.
Inocybe dulcamara (Pers.) P. Kumm.
Una buena cantidad de ejemplares brotando de manera dispersa, en la tierra removida al borde de un camino y bajo Pinus sylvestris.
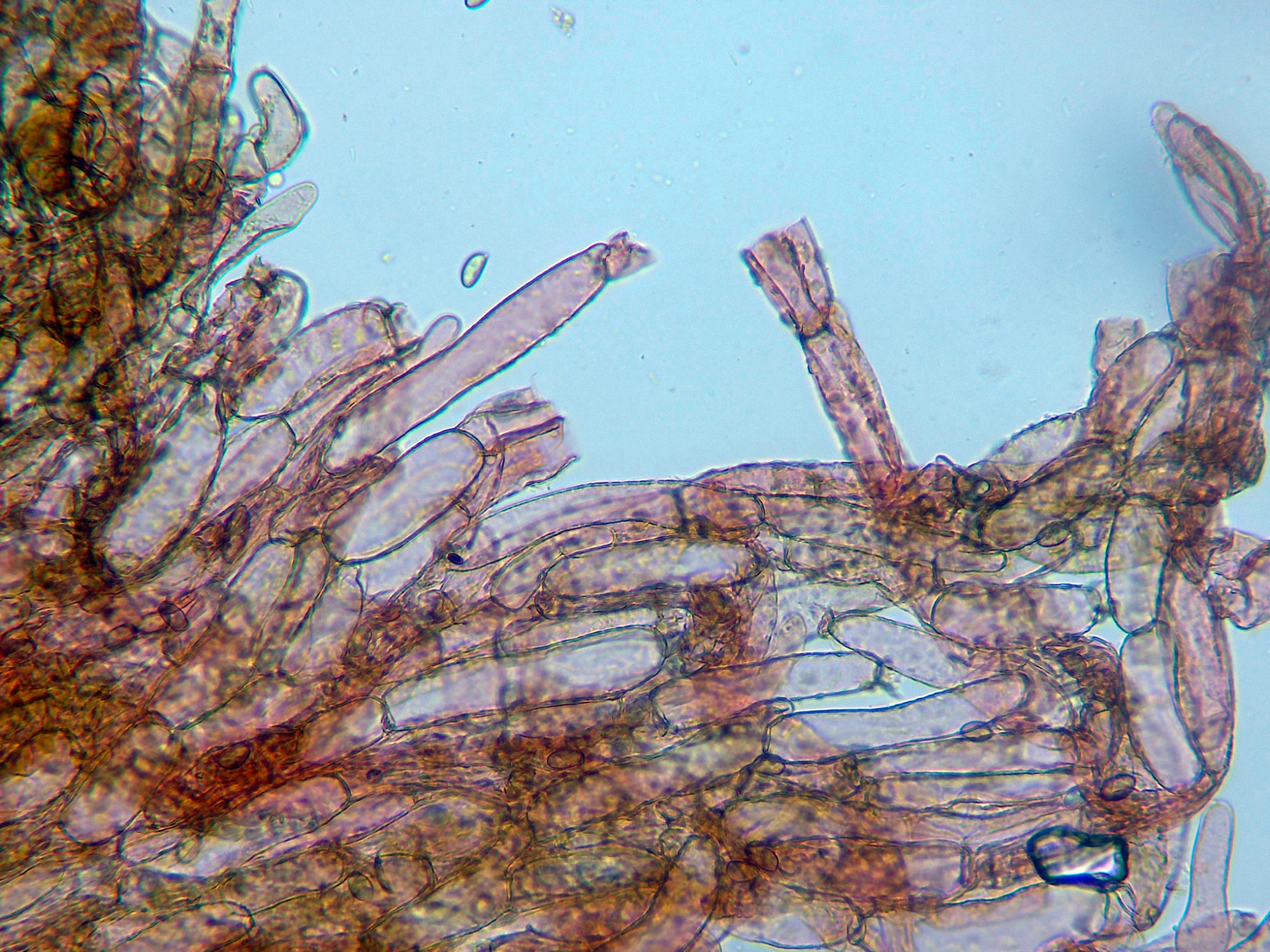
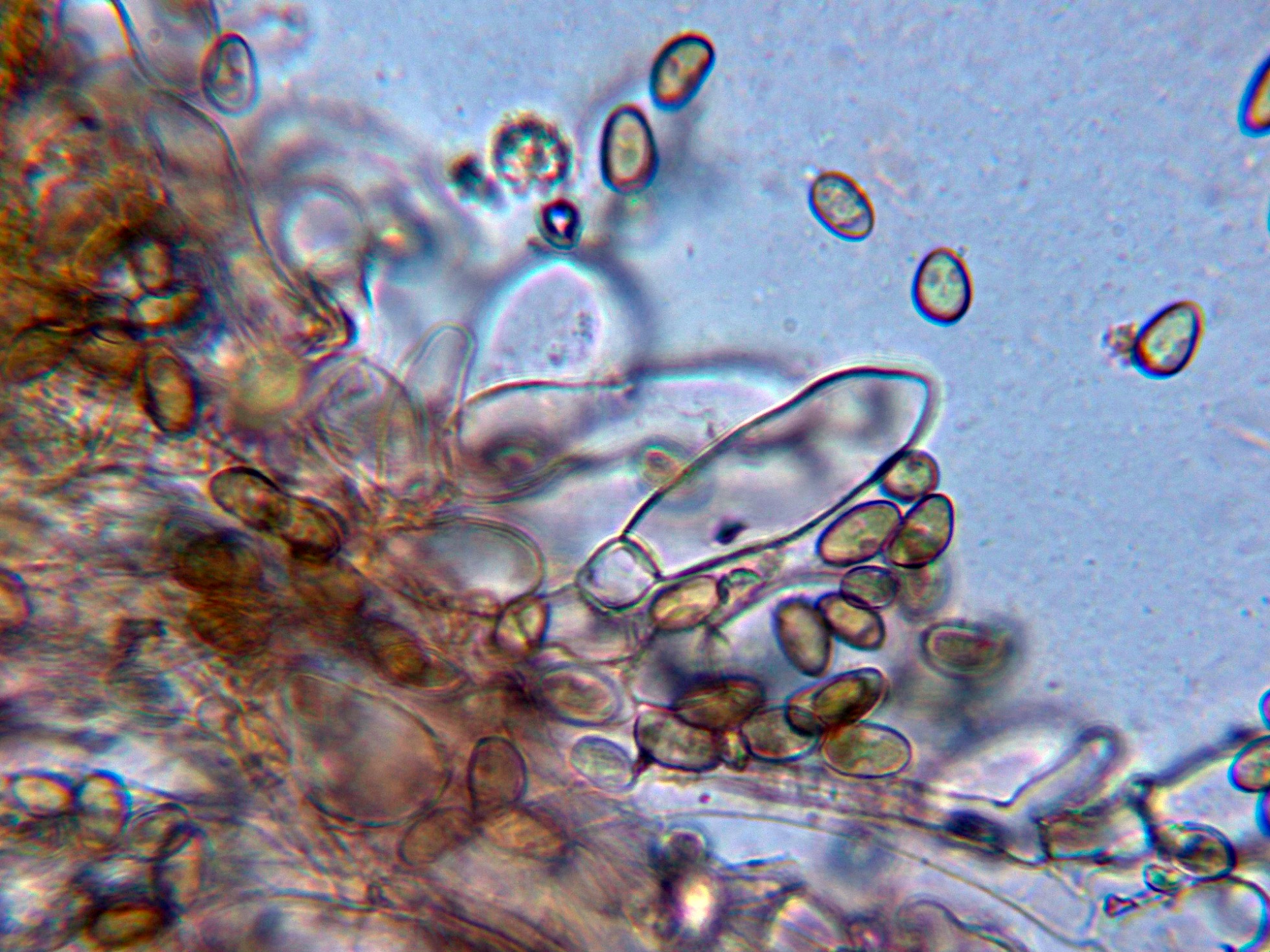
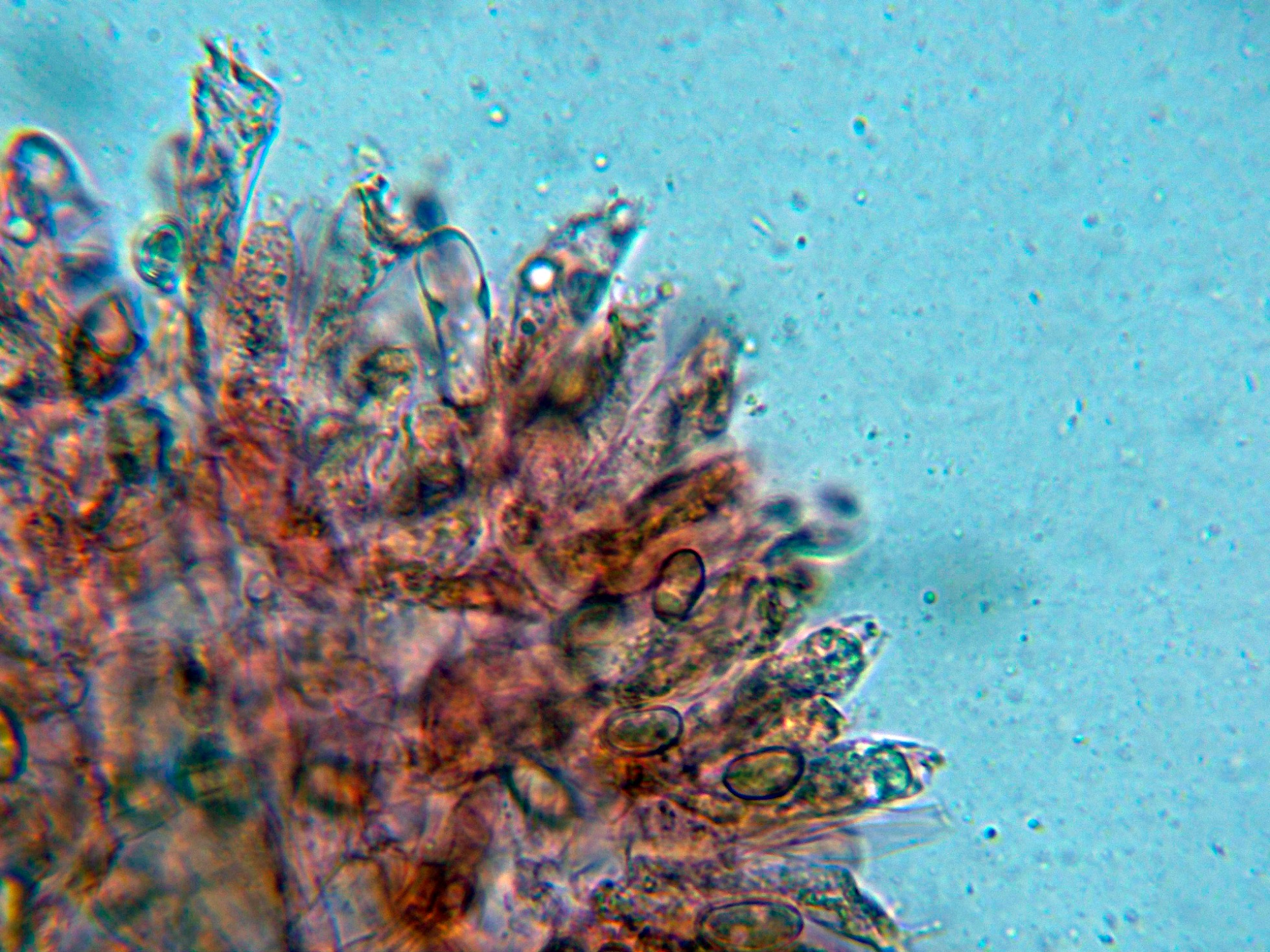
Pileipellis tipo trichoderma, con hifas filamentosas con una anchura de entre 7 y 13 micras, con pigmento amarillento en su interior y con algunas incrustaciones.
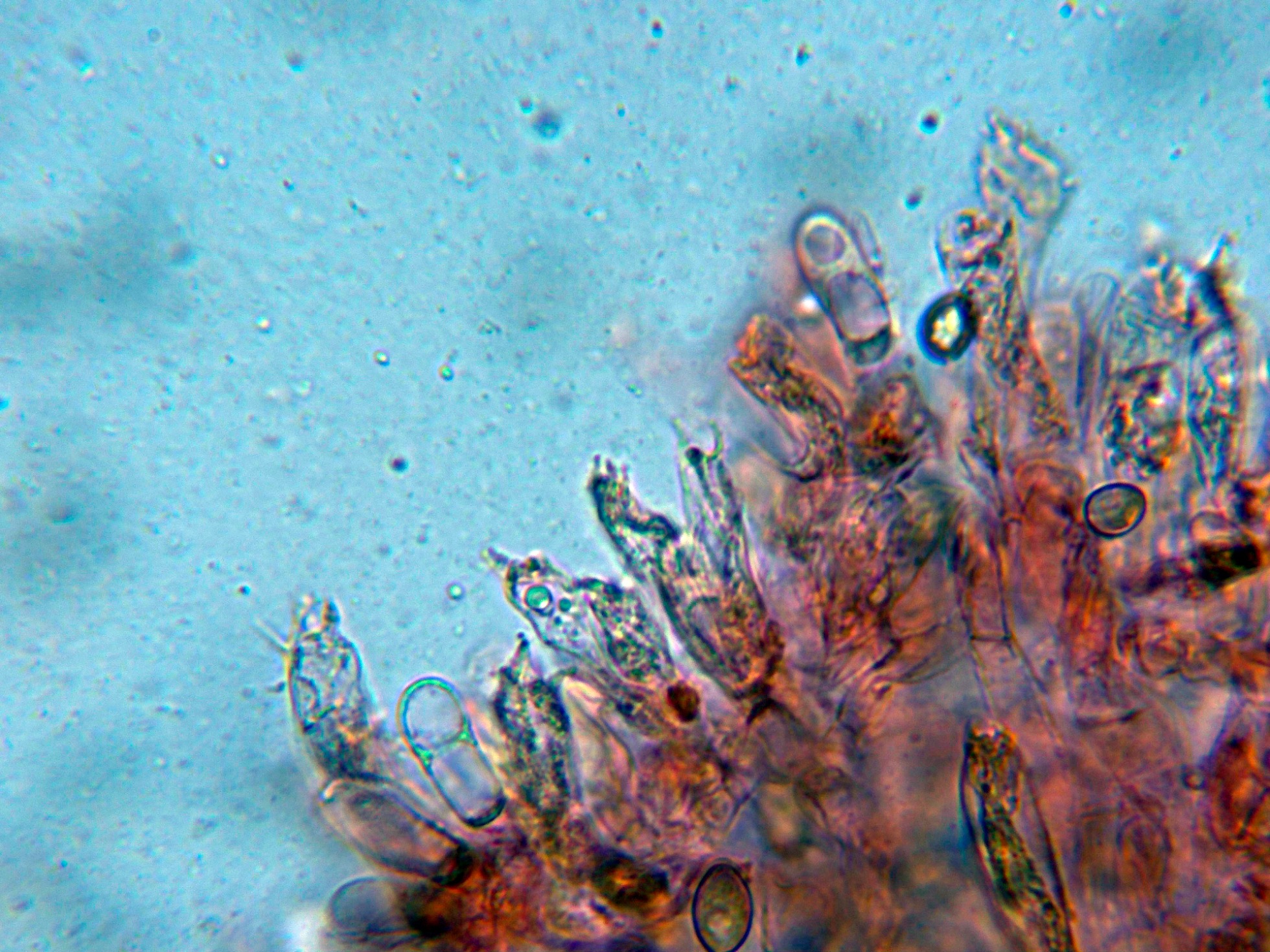
Las hifas de la pileipellis a 400 aumentos.
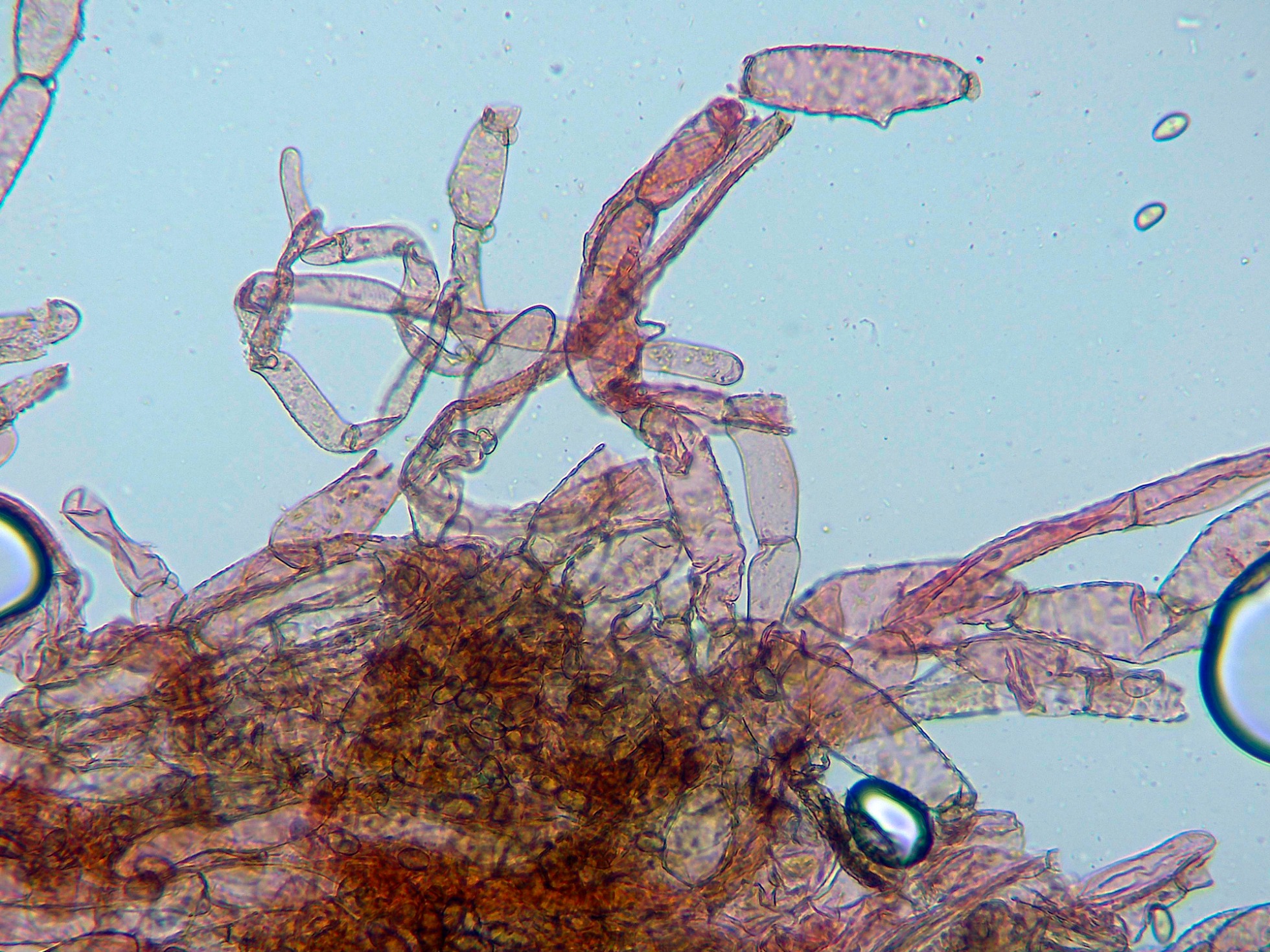
Las hifas de la suprapellis a 1000 aumentos, con fíbulas en los tabiques.
Hifas de la estipitipellis, hialinas y fibuladas.
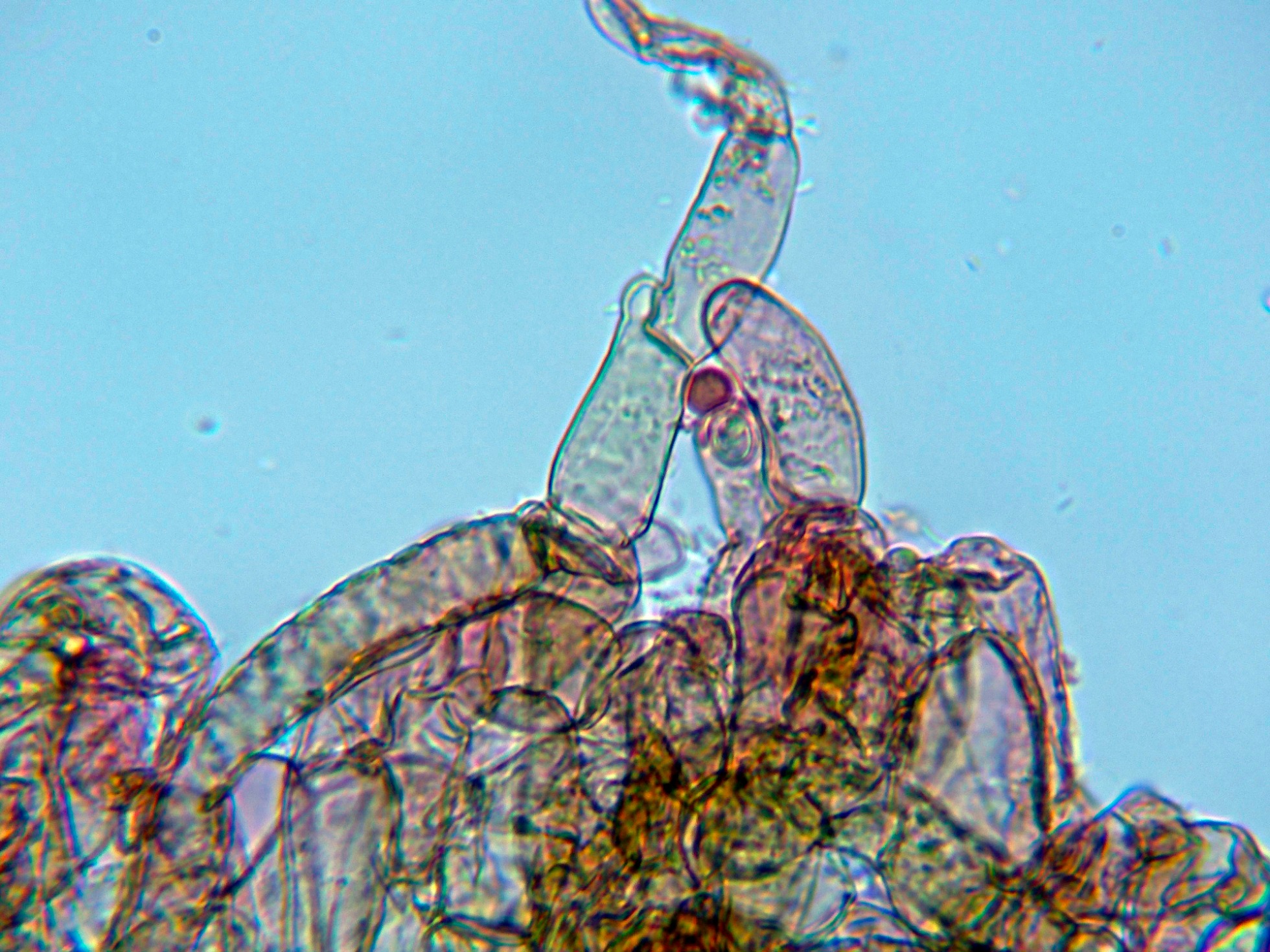
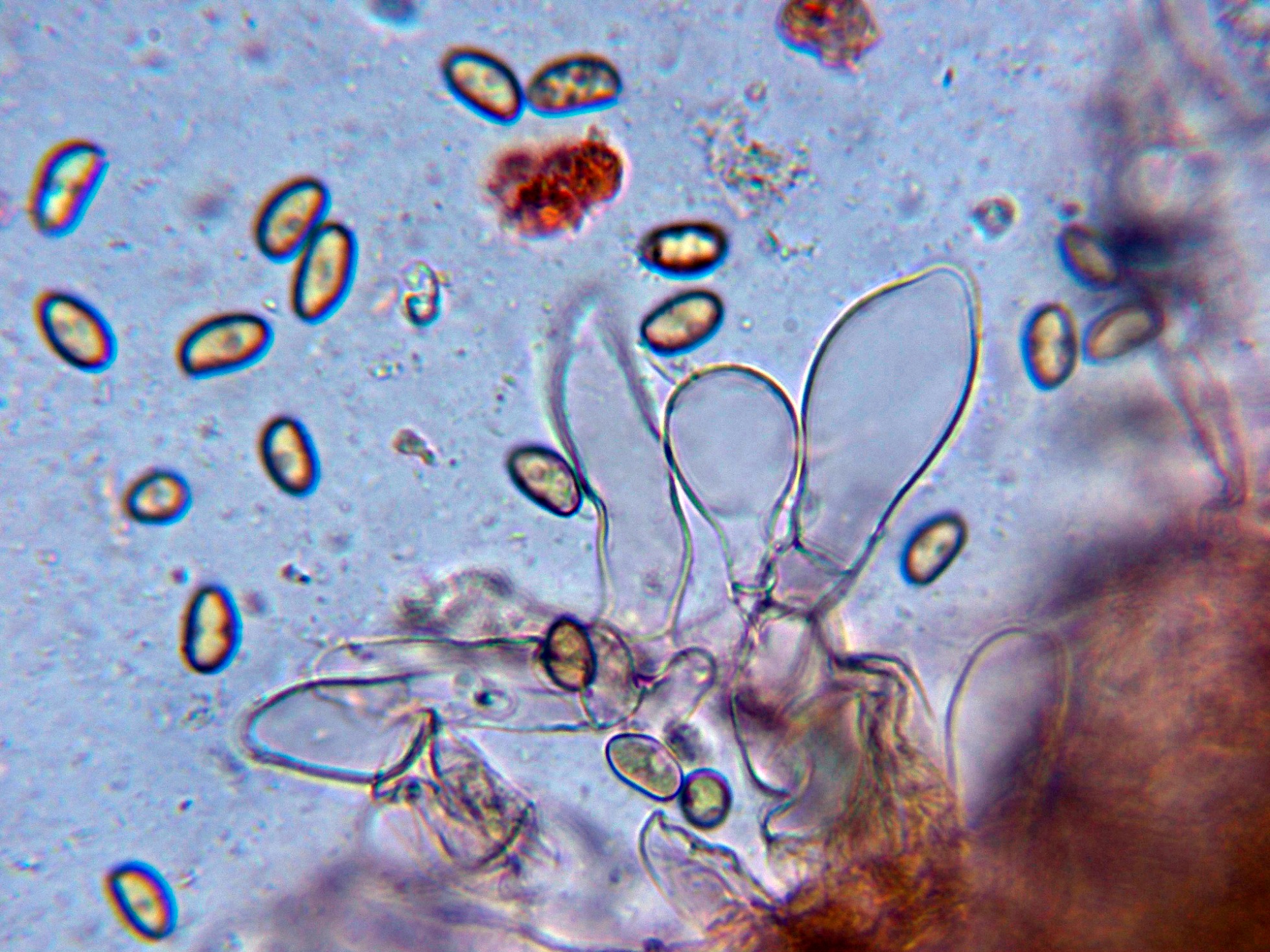
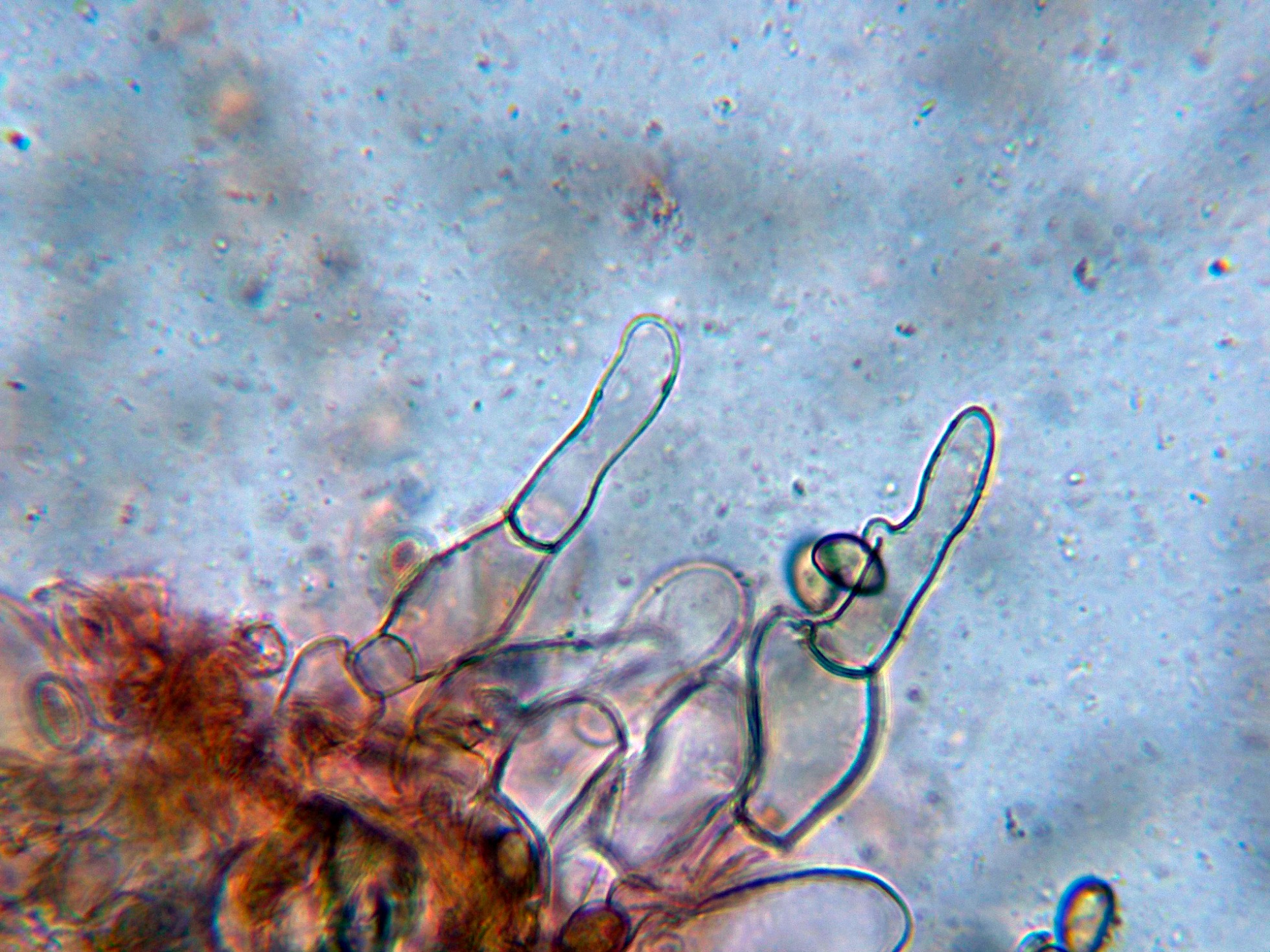
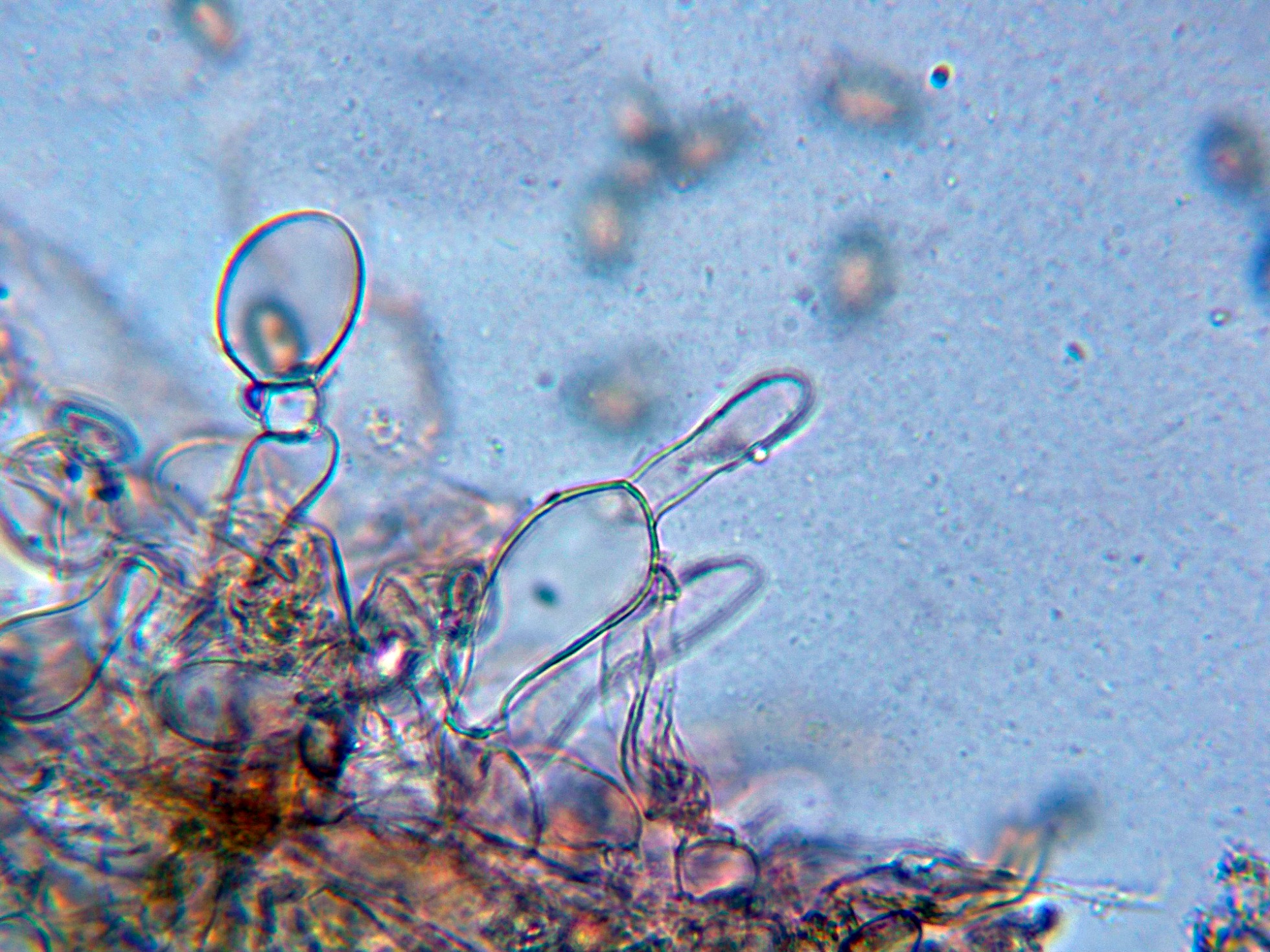
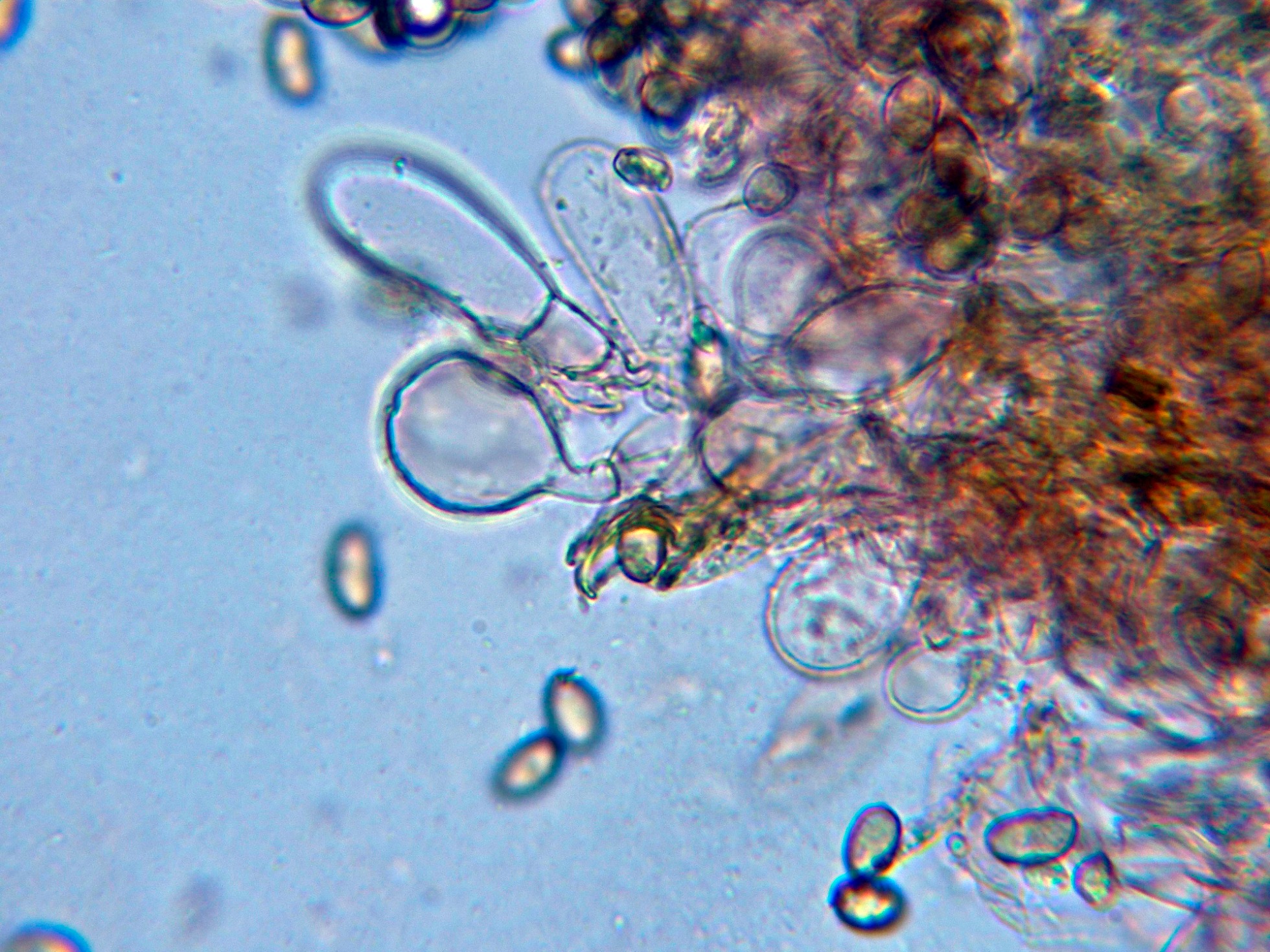
Queilocistidios tabicados con unas medidas de (19.8) 24.4 - 43.2 (45.6) × (6.7) 7.2 - 16.5 (16.8) µm.
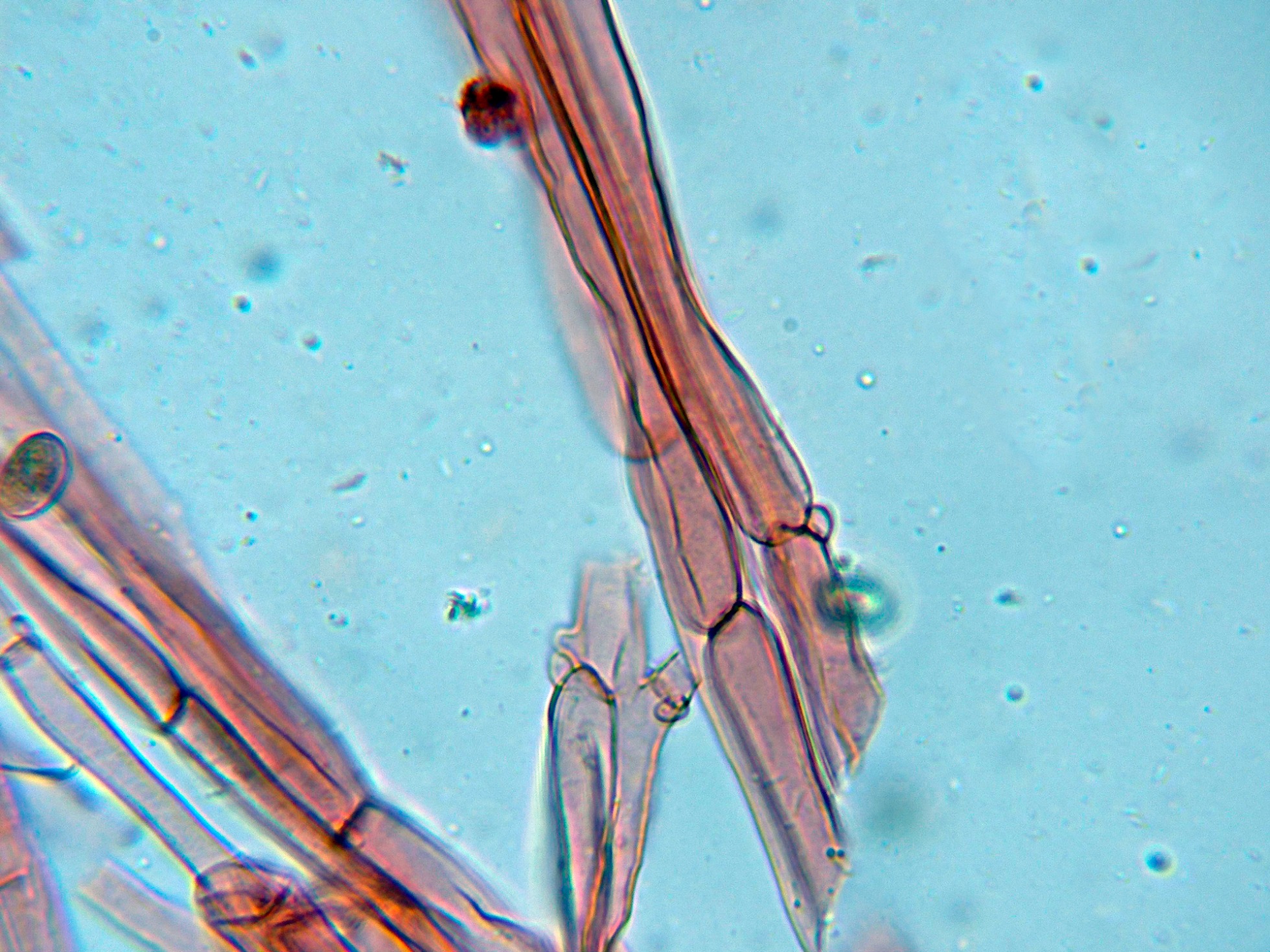
Basidios clavados, tetraspóricos y con fíbulas en su base.
Esporas en Reactivo de Melzer sin aparente reacción.
Esporas en KOH.
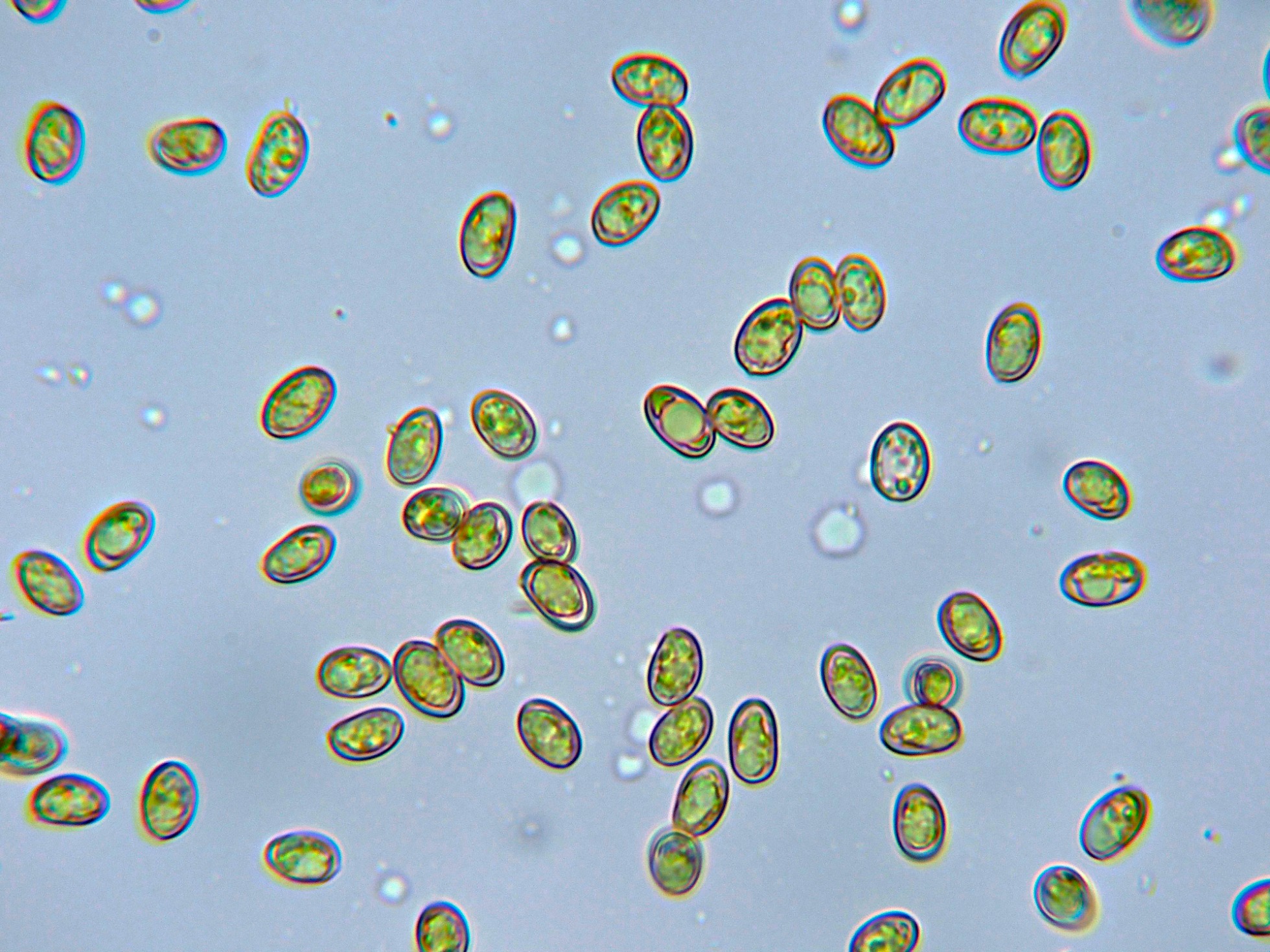
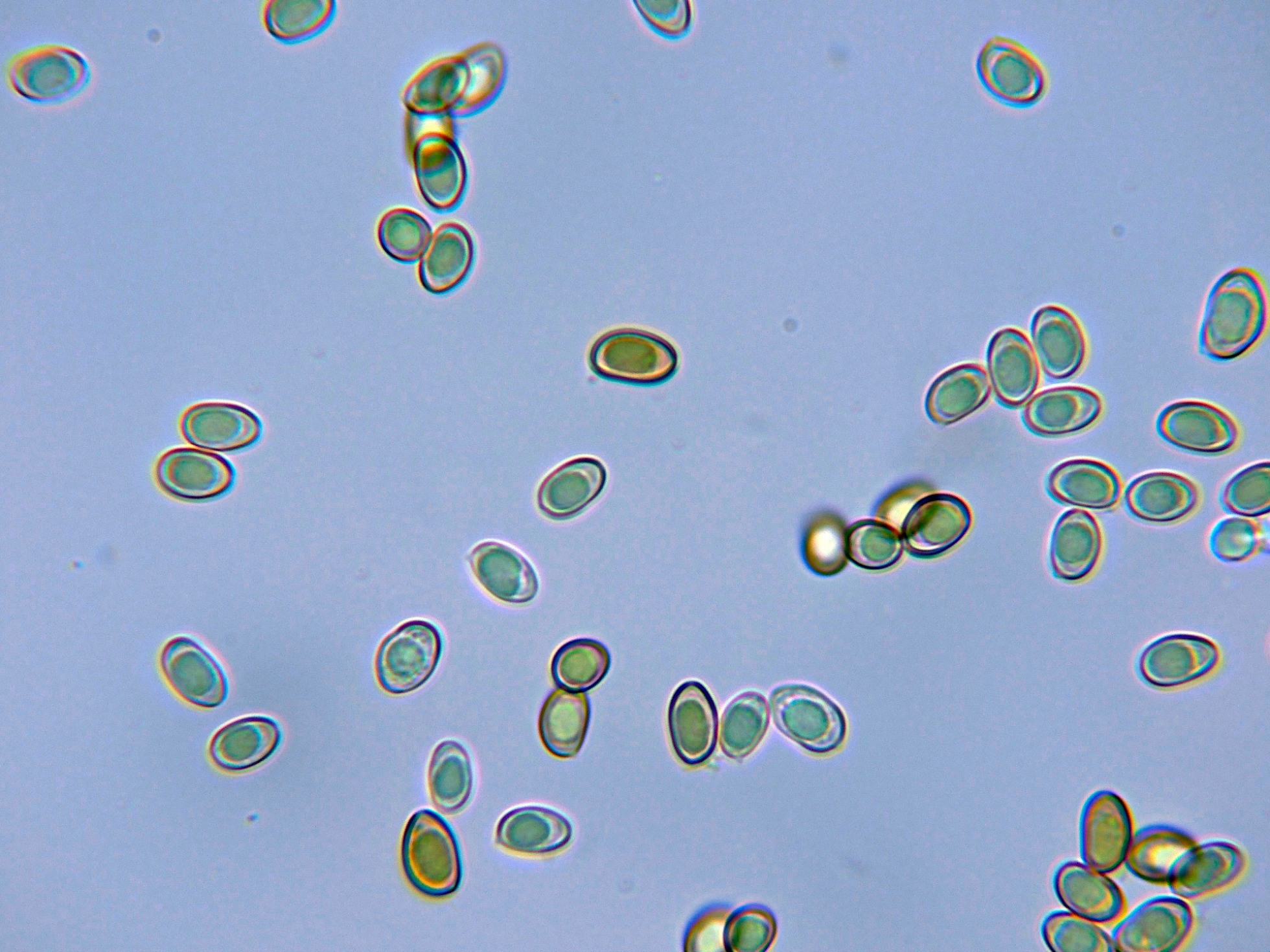
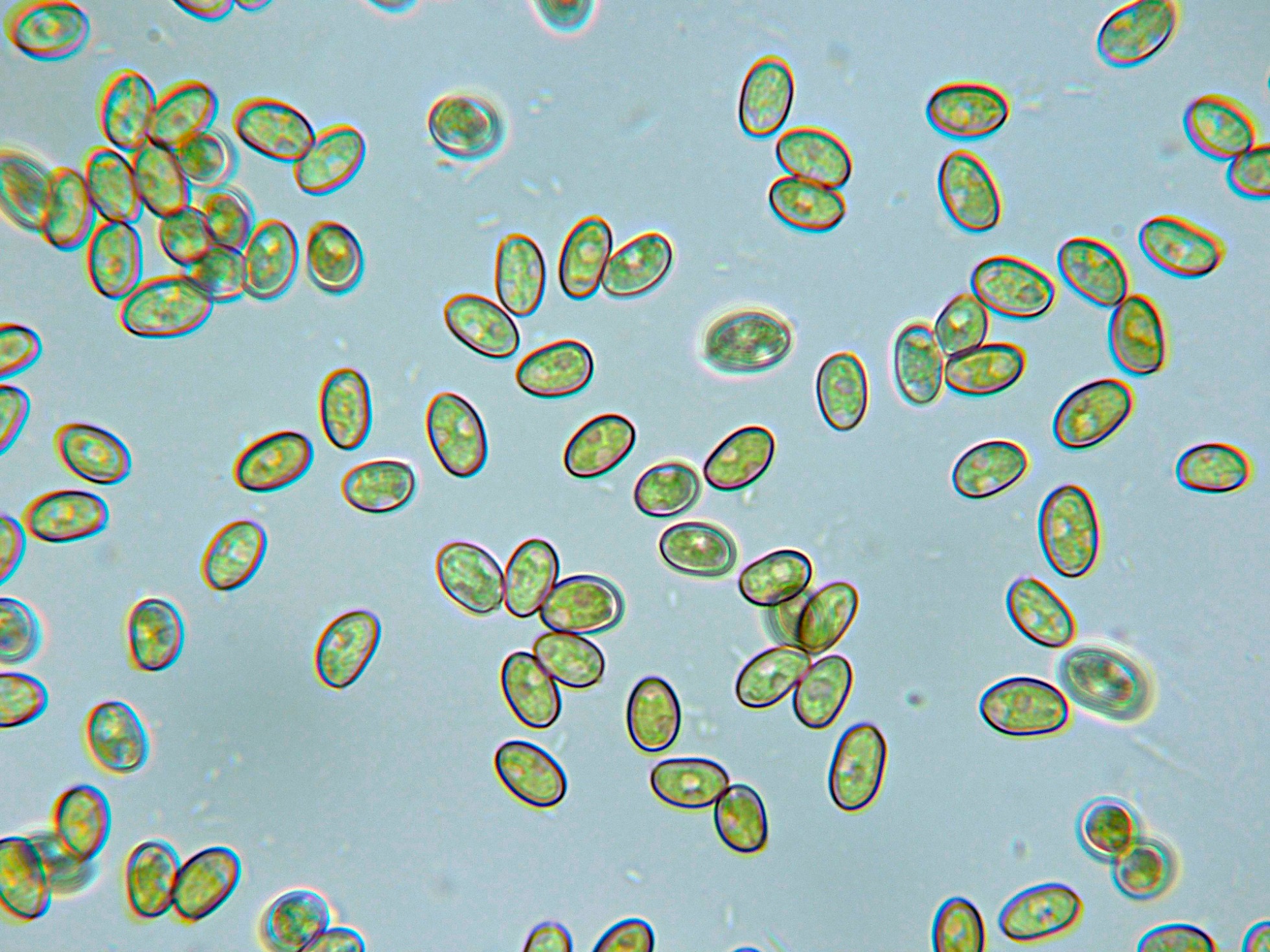
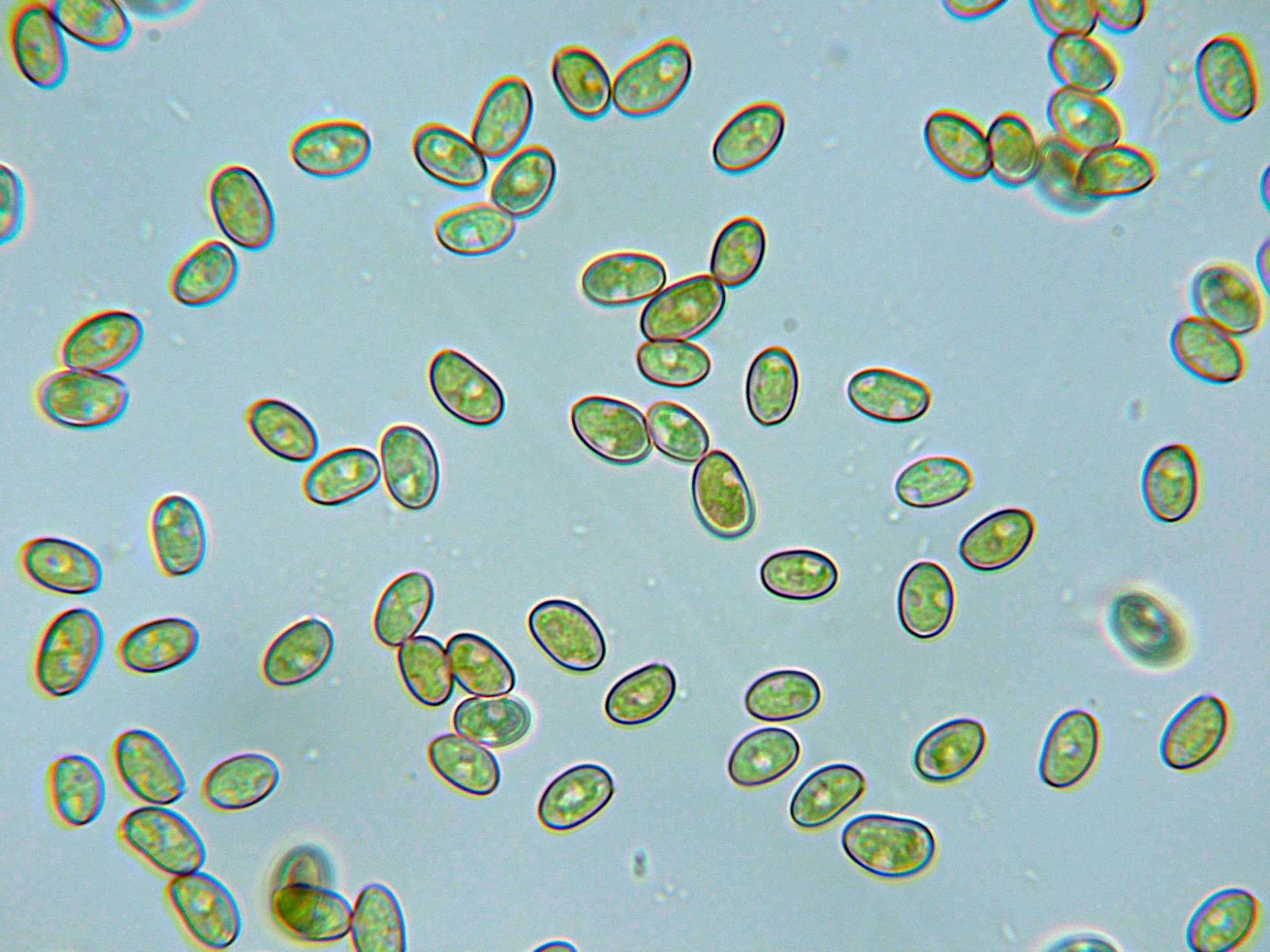
Esporas elipsoidales, pardo amarillentas al microscopio, con una discreta apícula lateral y unas medidas en agua de:
(7.8) 8.1 - 9.6 (10.7) × (5) 5.1 - 5.9 (6.3) µm
Q = (1.4) 1.5 - 1.7 (1.8) ; N = 40
Me = 8.8 × 5.5 µm ; Qe = 1.6
Las esporas en agua.
Aquí ya parecen encajar todas las piezas del puzzle, en Les Champignons de Suisse se indica una anchura para las hifas de la suprapellis de entre 6 y 12 µm. (en mi estudio de entre 7 y 13 µm.), unas medidas de los queilocistidios de 25-40 x 9-17 µm. (en este estudio de (19.8) 24.4 - 43.2 (45.6) × (6.7) 7.2 - 16.5 (16.8) µm.) y unas medidas esporales de 7.4 - 10.4 x 4.8 - 6.5 µm. ( en este estudio de (7.8) 8.1 - 9.6 (10.7) × (5) 5.1 - 5.9 (6.3) µm.) y un cociente Q de 1.3 - 1.9 (en este estudio de (1.4) 1.5 - 1.7 (1.8) con una media de Qe = 1.6.
Estos parámetros también parecen encajar con las Claves del género de Funga Nordica, donde se indican para la Inocybe dulcamara unas medidas de los queilocistidios de 30-50 x 10-20 μm. y unas medidas esporales de 8-11 x 5-6.5 μm,
Observaciones: A la Inocybe dulcamara se le suele asociar a terrenos arenosos como las dunas costeras, dejo un enlace de nuestra casa con un estudio realizado a la especie:
www.fungipedia.org/setas-informacion-y-c...kumm-1871.html#84266
No obstante todos los autores la sitúan además sobre suelos arenosos, al borde de caminos y bajo Pinus, como ha sido este el caso.
Saludos a tod@s.
El único hongo que tenía pendiente de identificar de mi recogida de muestras conjunta con Juan Andrés, Javi y Aitor, del pasado 30 de Octubre por tierras Burgalesas.
En el momento que saqué la microscopía no conseguí llegar a ninguna propuesta convincente y no tengo perdón, ya que si bien la especie no la había encontrado anteriormente, si que ya la había estudiado de unas muestras que me había traído un amigo allá por 2018 y además ya la había identificado correctamente en su momento, en esta ocasión para salir de dudas decidí recurrir al análisis molecular, la secuenciación ITS determinó que se trataba con una coincidencia del 99.58% de Mallocybe malençonii OQ133558. La Mallocybe malenconii = Inocybe malenconii ya creo haberla estudiado y en mi trabajo las esporas presentaban un cociente Q de (1.9) 2 - 2.4 (2.6), con una media de Qe = 2.2 y en esta ocasión el cociente Q estaba en Q = (1.4) 1.5 - 1.7 (1.8), con una media de Qe = 1.6. Según todas las Claves se indica un cociente Q para la Mallocybe malenconii superior a 2, con lo cuál era del todo imposible que en esta ocasión se tratara de este.
A nivel molecular se considera que dentro de la misma especie puede haber una diferencia de hasta un 0,5%, y aunque la coincidencia del 99.58% se encontraba dentro de este rango, queda pero que muy claro que hay en Genbank muchas secuencias mal identificadas, tal y como me ha tocado en esta ocasión.
Al disponer de tiempo y reabrir el tema pronto he dado con la solución.
Inocybe dulcamara (Pers.) P. Kumm.
Una buena cantidad de ejemplares brotando de manera dispersa, en la tierra removida al borde de un camino y bajo Pinus sylvestris.
Pileipellis tipo trichoderma, con hifas filamentosas con una anchura de entre 7 y 13 micras, con pigmento amarillento en su interior y con algunas incrustaciones.
Las hifas de la pileipellis a 400 aumentos.
Las hifas de la suprapellis a 1000 aumentos, con fíbulas en los tabiques.
Hifas de la estipitipellis, hialinas y fibuladas.
Queilocistidios tabicados con unas medidas de (19.8) 24.4 - 43.2 (45.6) × (6.7) 7.2 - 16.5 (16.8) µm.
Basidios clavados, tetraspóricos y con fíbulas en su base.
Esporas en Reactivo de Melzer sin aparente reacción.
Esporas en KOH.
Esporas elipsoidales, pardo amarillentas al microscopio, con una discreta apícula lateral y unas medidas en agua de:
(7.8) 8.1 - 9.6 (10.7) × (5) 5.1 - 5.9 (6.3) µm
Q = (1.4) 1.5 - 1.7 (1.8) ; N = 40
Me = 8.8 × 5.5 µm ; Qe = 1.6
Las esporas en agua.
Aquí ya parecen encajar todas las piezas del puzzle, en Les Champignons de Suisse se indica una anchura para las hifas de la suprapellis de entre 6 y 12 µm. (en mi estudio de entre 7 y 13 µm.), unas medidas de los queilocistidios de 25-40 x 9-17 µm. (en este estudio de (19.8) 24.4 - 43.2 (45.6) × (6.7) 7.2 - 16.5 (16.8) µm.) y unas medidas esporales de 7.4 - 10.4 x 4.8 - 6.5 µm. ( en este estudio de (7.8) 8.1 - 9.6 (10.7) × (5) 5.1 - 5.9 (6.3) µm.) y un cociente Q de 1.3 - 1.9 (en este estudio de (1.4) 1.5 - 1.7 (1.8) con una media de Qe = 1.6.
Estos parámetros también parecen encajar con las Claves del género de Funga Nordica, donde se indican para la Inocybe dulcamara unas medidas de los queilocistidios de 30-50 x 10-20 μm. y unas medidas esporales de 8-11 x 5-6.5 μm,
Observaciones: A la Inocybe dulcamara se le suele asociar a terrenos arenosos como las dunas costeras, dejo un enlace de nuestra casa con un estudio realizado a la especie:
www.fungipedia.org/setas-informacion-y-c...kumm-1871.html#84266
No obstante todos los autores la sitúan además sobre suelos arenosos, al borde de caminos y bajo Pinus, como ha sido este el caso.
Saludos a tod@s.
Adjuntos:
El siguiente usuario dijo gracias: Juan Andrés Román, Juan
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
- Javi Calvo Pérez
-

- Fuera de línea
- Spammer
-

Menos
Más
- Mensajes: 6980
- Gracias recibidas: 2922
2 años 1 mes antes #108663
por Javi Calvo Pérez
Respuesta de Javi Calvo Pérez sobre el tema Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
Muy bueno Josep, el que la sigue la consigue, mira que la conocía de las dunas, la diferencia en el q esporal con respecto a Mallocybe malenconii es clara y la micro no deja lugar a dudas. Curioso que la secuencia fallará, acaso no está secuenciada Inocybe dulcamara? Saludos desde el frío invierno vasco
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
- JOSEP
-
 Autor del tema
Autor del tema
- Fuera de línea
- Moderador
-

Menos
Más
- Mensajes: 8430
- Gracias recibidas: 8310
2 años 1 mes antes #108669
por JOSEP
Respuesta de JOSEP sobre el tema Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
Hola Javi.
No te quepa ninguna duda de que la Inocybe dulcamara está sobradamente secuenciada, el resultado que se consigue con la secuenciación es solo que se me transcriba los códigos de las secuencias que más se parecen a la mía, en este caso en cuanto a la fracción ITS lo más parecido a la mía era una que identificaron erróneamente como Mallocybe malenconii, el problema reside que hay muchas secuencias en Genbank mal identificadas, como parece ser que ha sido este el caso, en casos como este la falta de datos de buena calidad en Genbank es el factor limitante, y al final tengo que ser yo mismo el que tiene que solucionar los problemas taxonómicos. Además puede suceder que en muestras con un ITS 100% idéntico sean diferentes especies (ya me sucedió con anterioridad con la Hohenbuehelia atrocoerulea que coincidía con 4), con lo que se tiene que recurrir a otros genes como LSU, rpb2, tef1, etc., y aún así tampoco se puede garantizar que se puedan separar adecuadamente, esto solo se conseguiría con el árbol filogenético y una comparativa entre ellas.
Afortunadamente al final he podido dar con su identidad correcta.
Un abrazo
No te quepa ninguna duda de que la Inocybe dulcamara está sobradamente secuenciada, el resultado que se consigue con la secuenciación es solo que se me transcriba los códigos de las secuencias que más se parecen a la mía, en este caso en cuanto a la fracción ITS lo más parecido a la mía era una que identificaron erróneamente como Mallocybe malenconii, el problema reside que hay muchas secuencias en Genbank mal identificadas, como parece ser que ha sido este el caso, en casos como este la falta de datos de buena calidad en Genbank es el factor limitante, y al final tengo que ser yo mismo el que tiene que solucionar los problemas taxonómicos. Además puede suceder que en muestras con un ITS 100% idéntico sean diferentes especies (ya me sucedió con anterioridad con la Hohenbuehelia atrocoerulea que coincidía con 4), con lo que se tiene que recurrir a otros genes como LSU, rpb2, tef1, etc., y aún así tampoco se puede garantizar que se puedan separar adecuadamente, esto solo se conseguiría con el árbol filogenético y una comparativa entre ellas.
Afortunadamente al final he podido dar con su identidad correcta.
Un abrazo
El siguiente usuario dijo gracias: Juan Andrés Román, Juan
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
- Juan
-

- Fuera de línea
- Moderador
-

Menos
Más
- Mensajes: 553
- Gracias recibidas: 608
2 años 1 mes antes #108673
por Juan
Respuesta de Juan sobre el tema Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
Un gran trabajo como siempre Josep e información nueva que no conocía, gracias! 
El siguiente usuario dijo gracias: Juan Andrés Román, JOSEP
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
- Foro
- Foros sobre Micología de fungipedia
- Microscopía
- Enigma resuelto: Inocybe dulcamara (Pers.) P. Kumm.
Tiempo de carga de la página: 0.176 segundos


















Foro de micología