 Una nueva especie de Exarmidium. Microscopía.
Una nueva especie de Exarmidium. Microscopía.
- JOSEP
-
 Autor del tema
Autor del tema
- Fuera de línea
- Moderador
-

Menos
Más
- Mensajes: 8417
- Gracias recibidas: 8310
8 meses 2 semanas antes #110069
por JOSEP
Una nueva especie de Exarmidium. Microscopía. Publicado por JOSEP
Hola a tod@s.
Un Exarmidium fotografiado el 1 de mayo de 2023, en un punto cercano a la localidad de Coll de Nargó (Lleida).
Unos pequeños apotecios brotando de manera dispersa pero abundante sobre una tabla de madera ya elaborada y trabajada y además bastante degradada de probablemente planifolio.
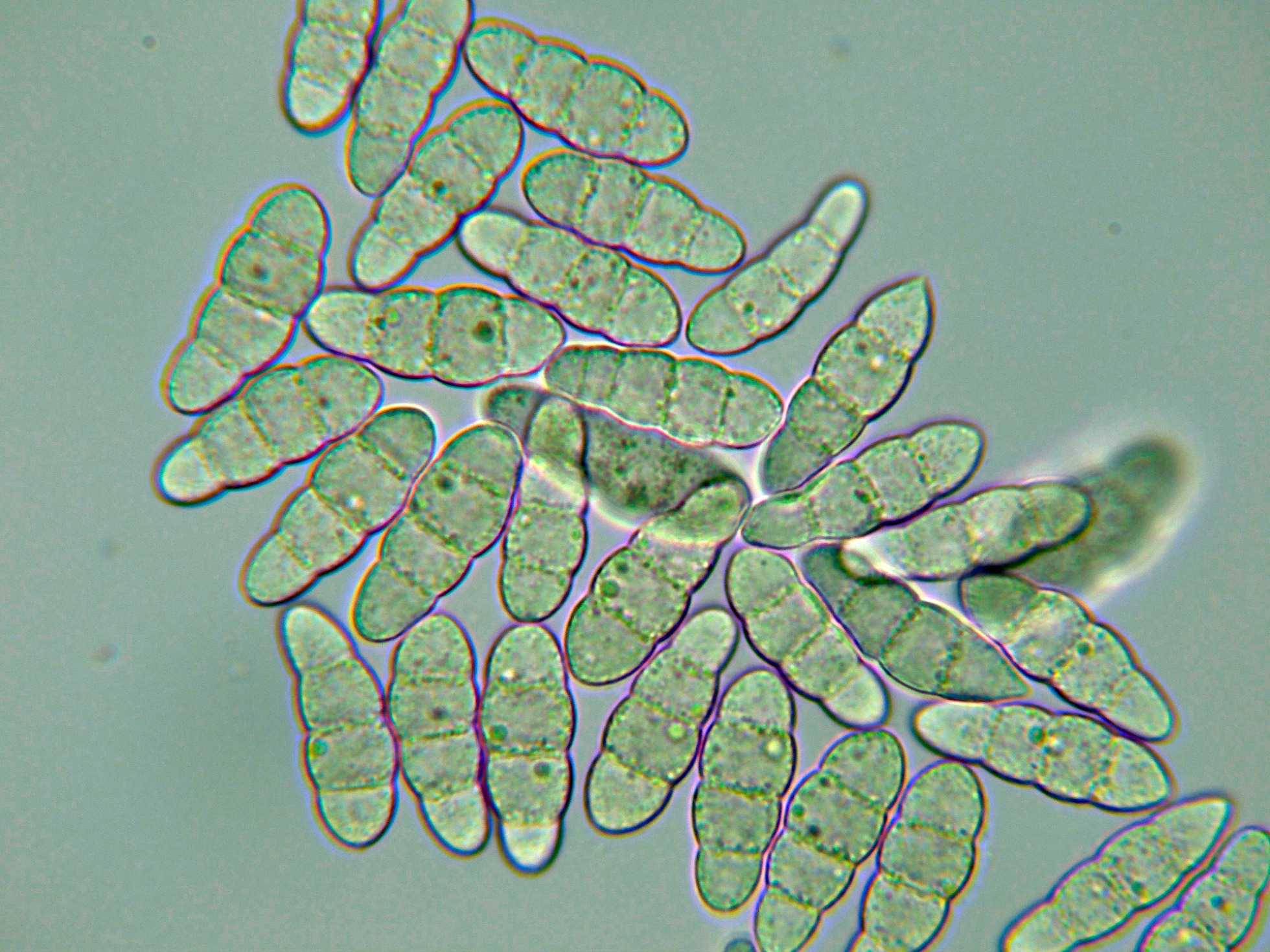
Los ascomas a 20 aumentos:
Otra imágen de los ascomas a 20 aumentos con la escala:
Los ascomas a 40 aumentos:
Estos ascomas a 40 aumentos con sus medidas:
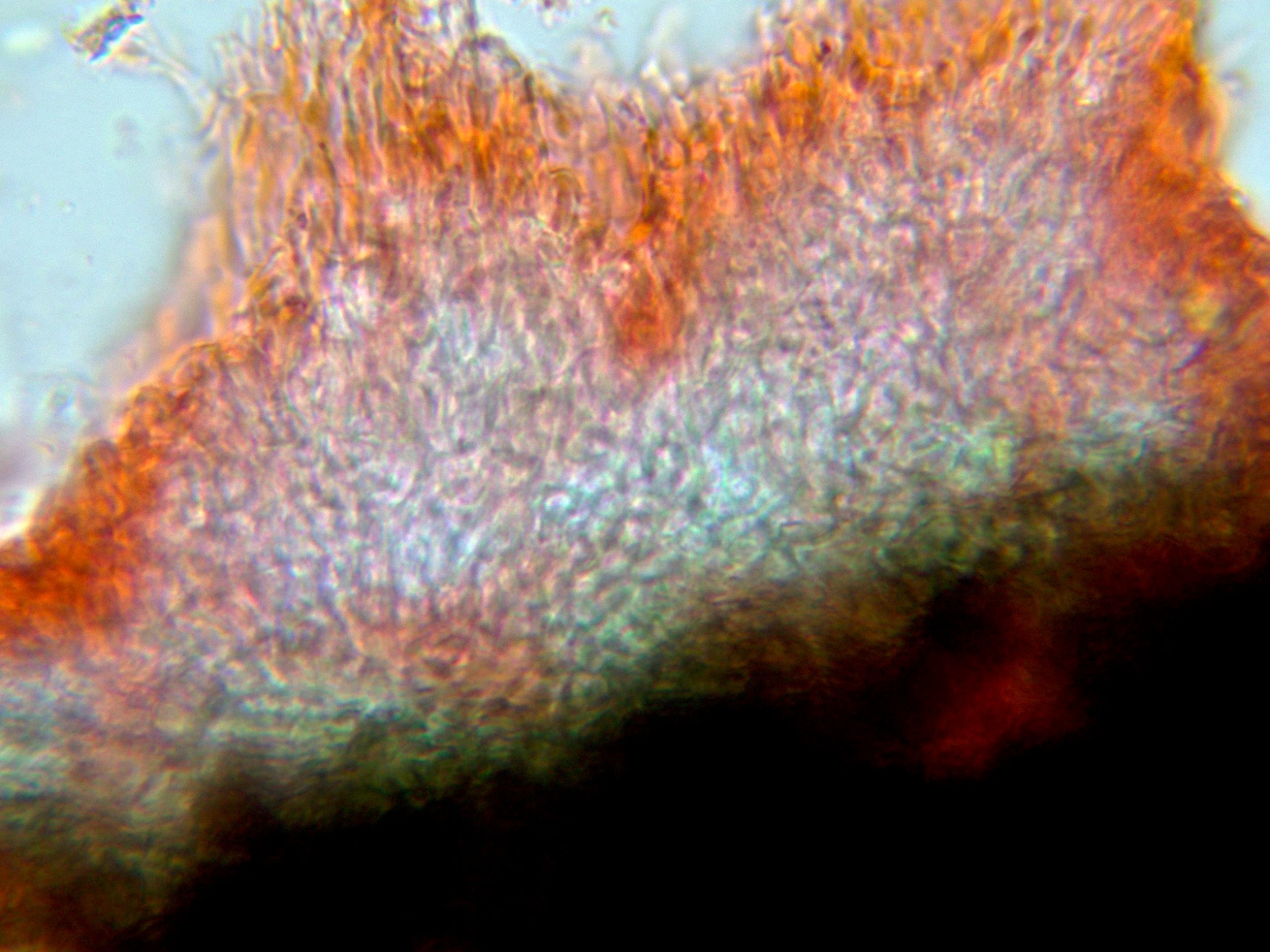
La sección de los ascomas a 40 aumentos:
La sección de un ascoma con sus medidas:
Las hifas pigmentadas de la pared del peritecio:
El himenio a 400 aumentos en solución de Rojo Congo diluido:
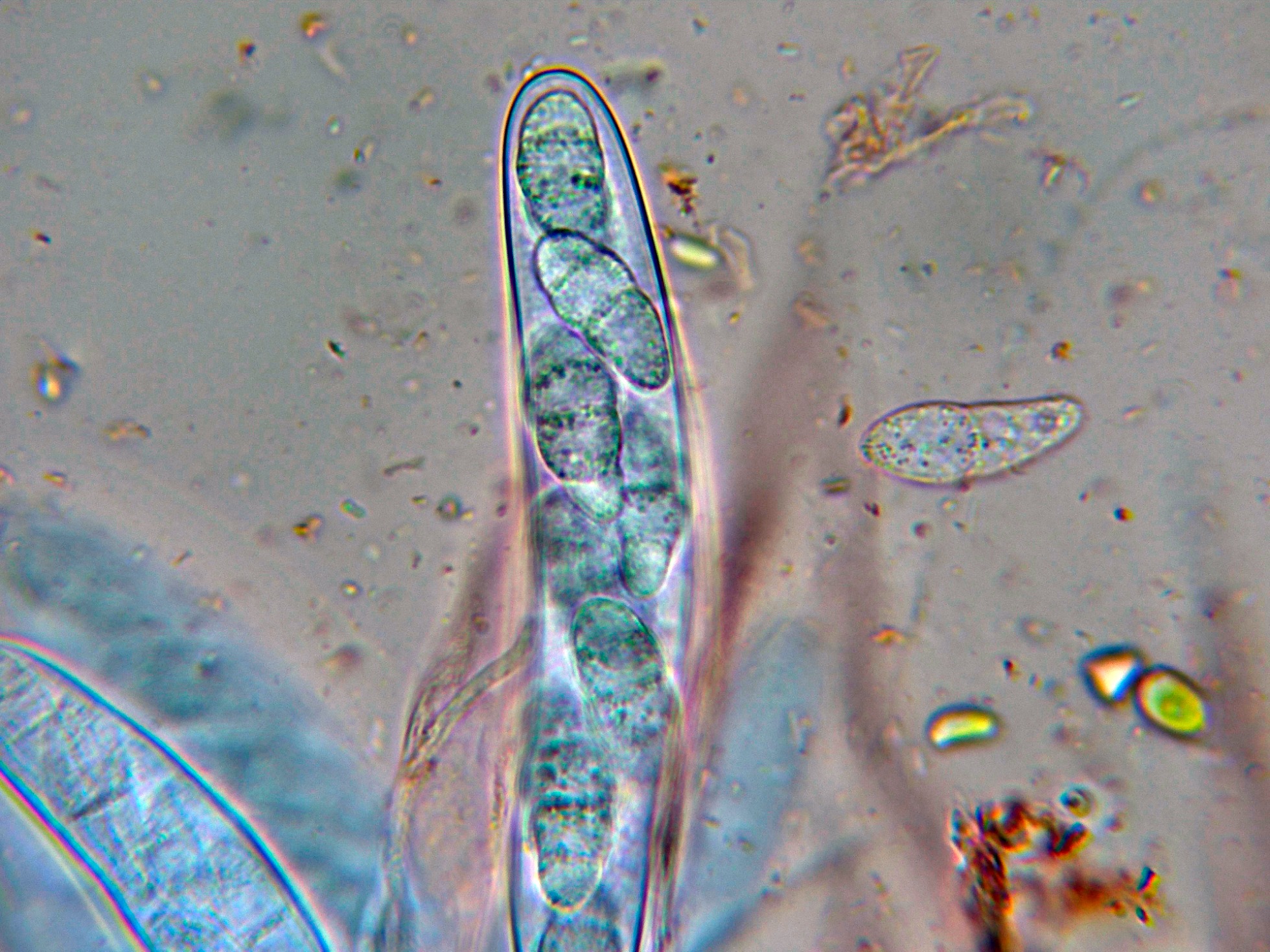
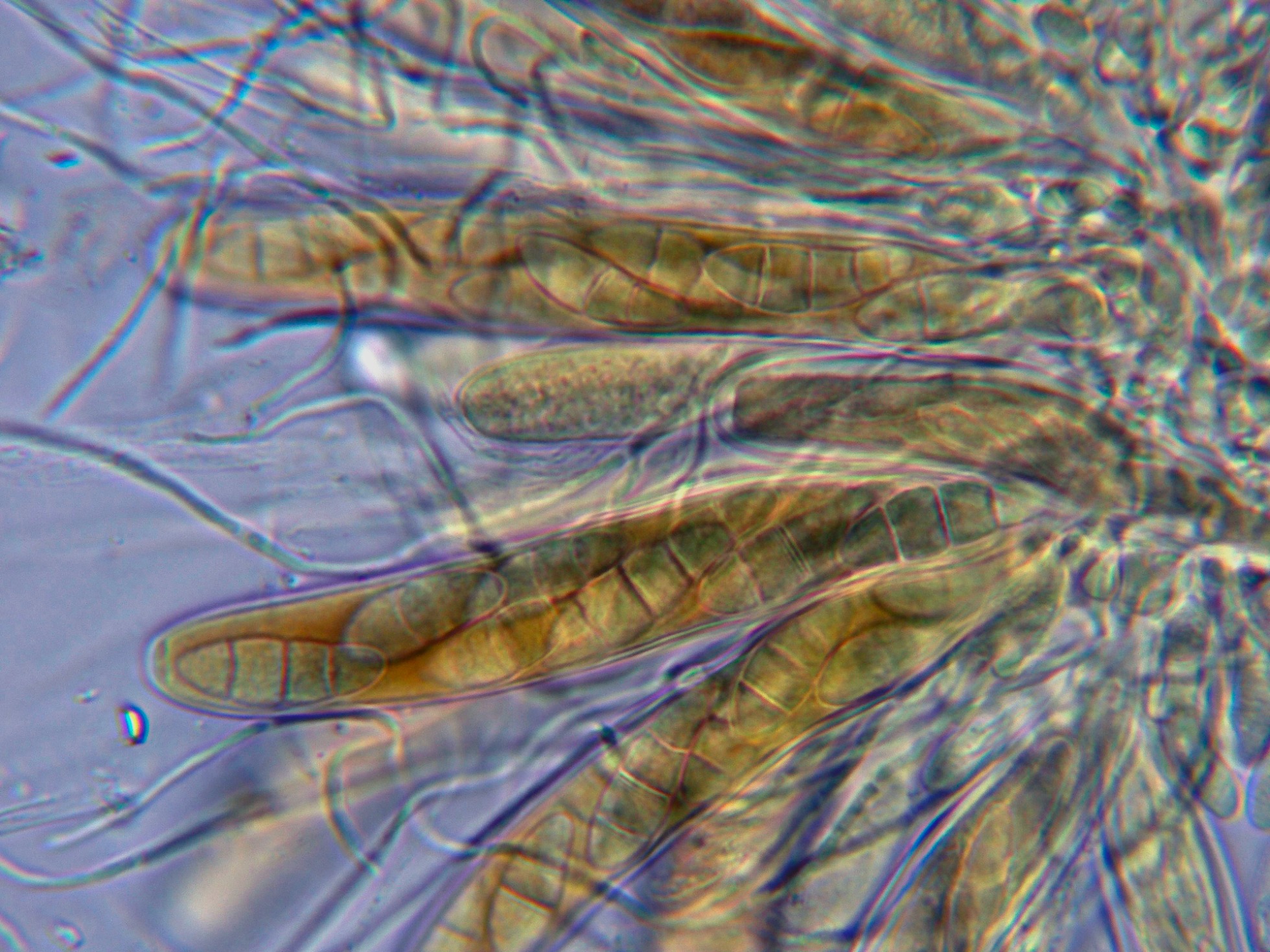
Los ascos en solución de Rojo Congo a 1000 aumentos con unas medidas de (81.4) 82.6 - 108.1 (121) × (13.9) 14.7 - 17.3 (19) µm.
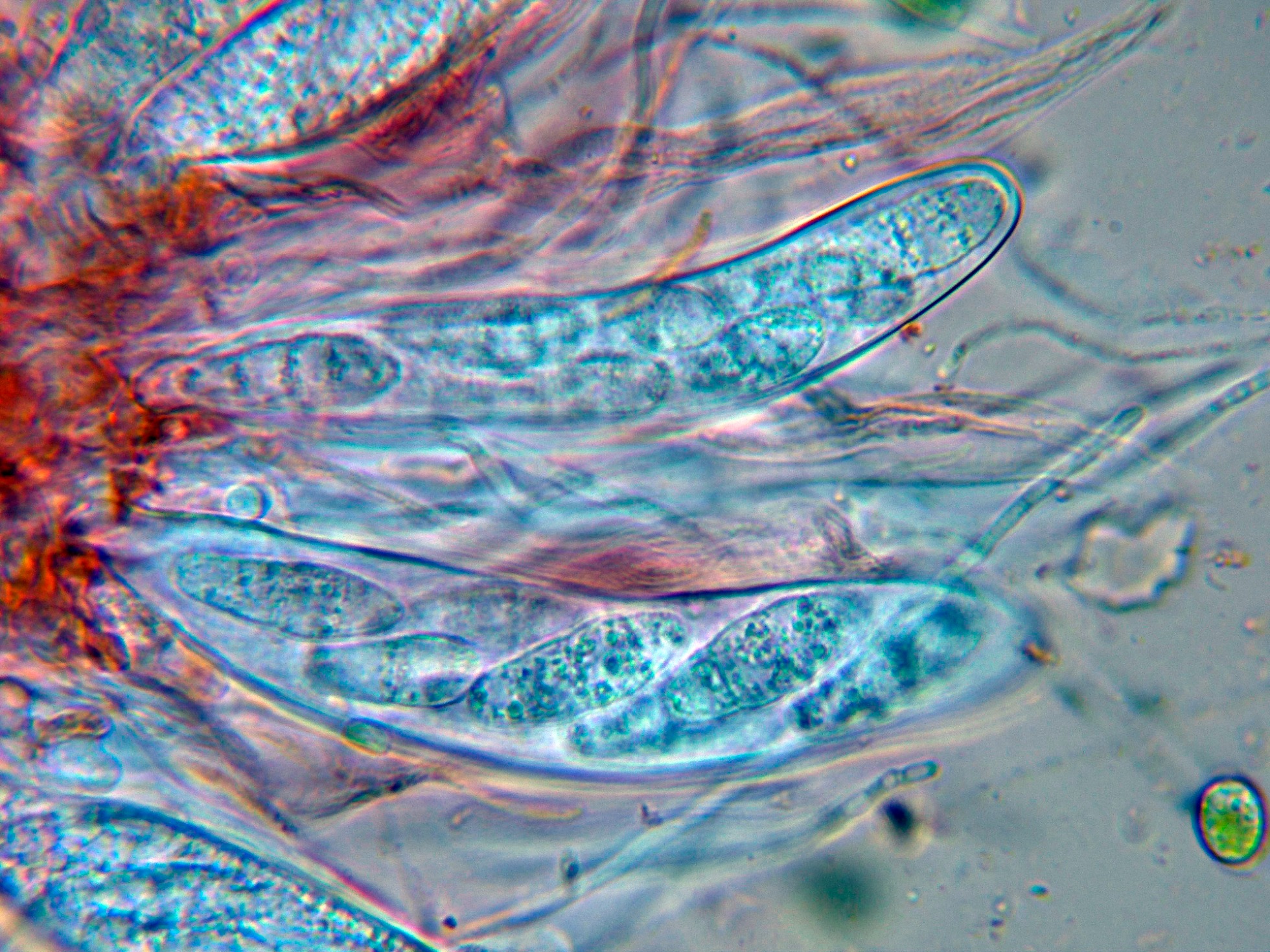
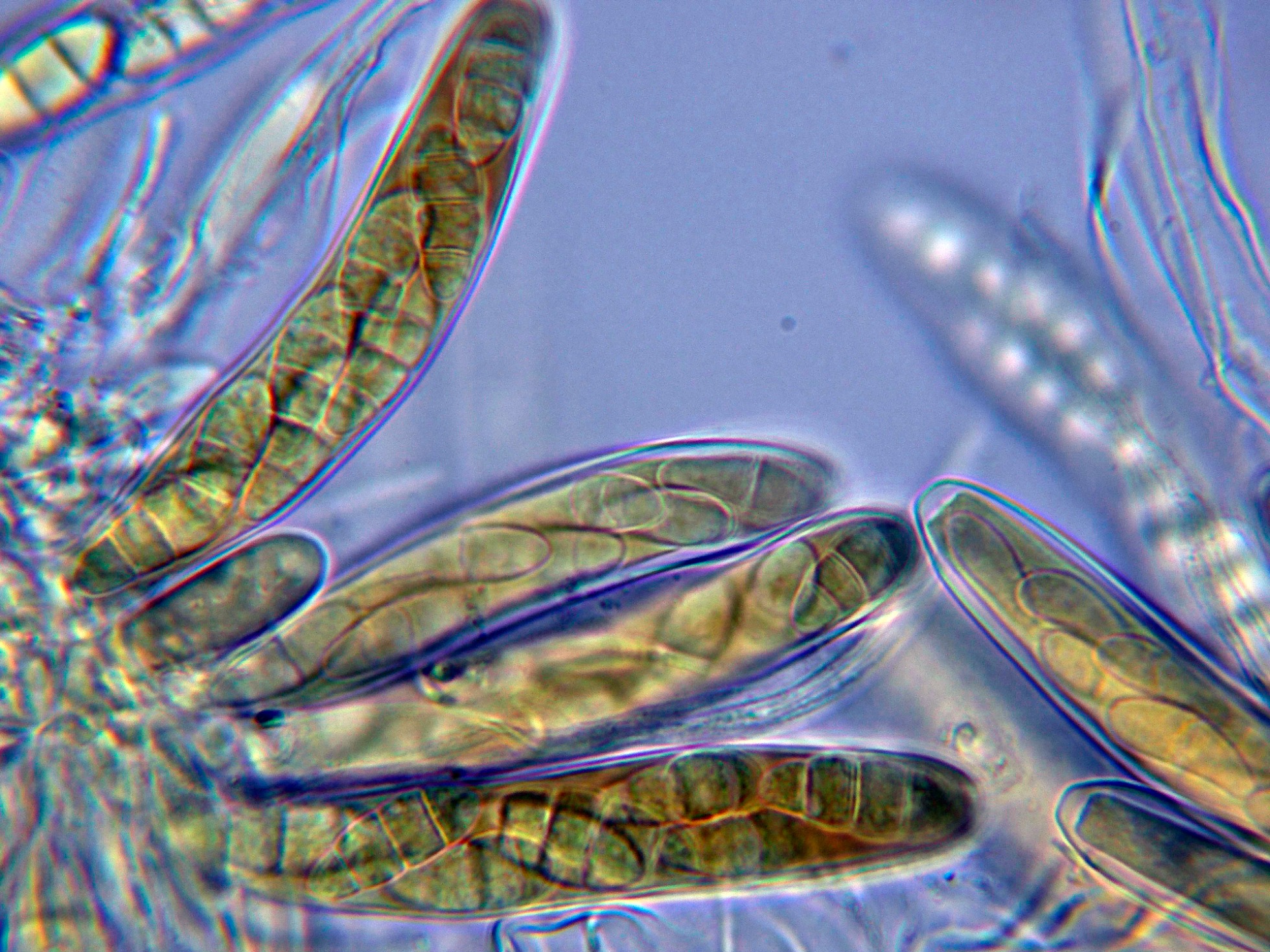
Los ascos en Reactivo de Melzer, con reacción débilmente dextrinoide:
Al utilizar Lugol, la reacción dextrinoide de los ascos se hace ya más evidente:
Los ascos en Lugol a 400 aumentos:
Los ascos en Lugol a 1000 aumentos:
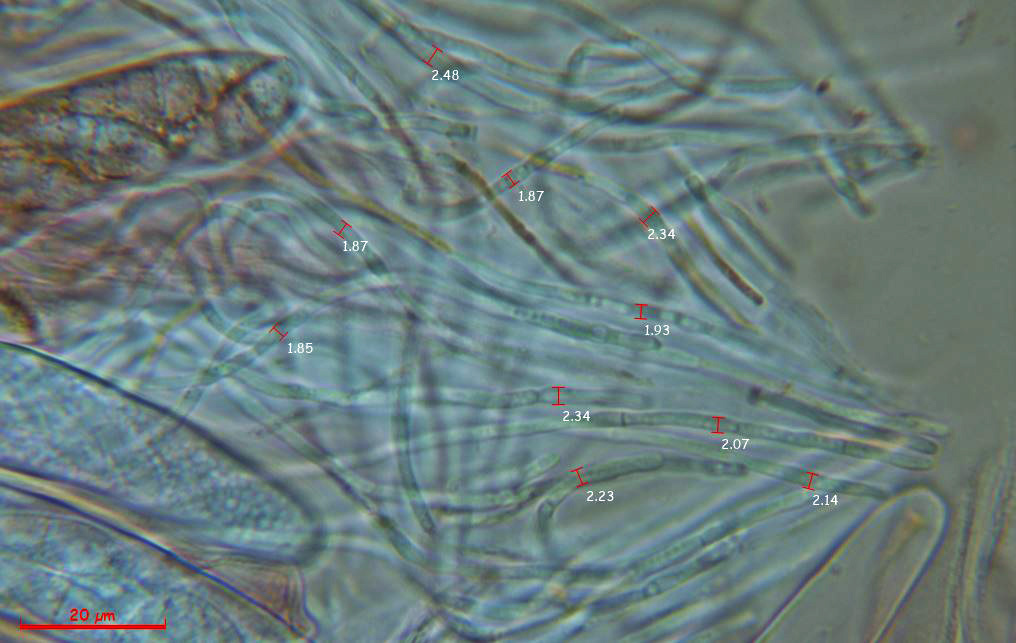
Materia interascal a modo de paráfisis, muy densa, filiforme, con septos y una anchura de entre 1,85 a 2,45 µm.
La materia interascal con su anchura:
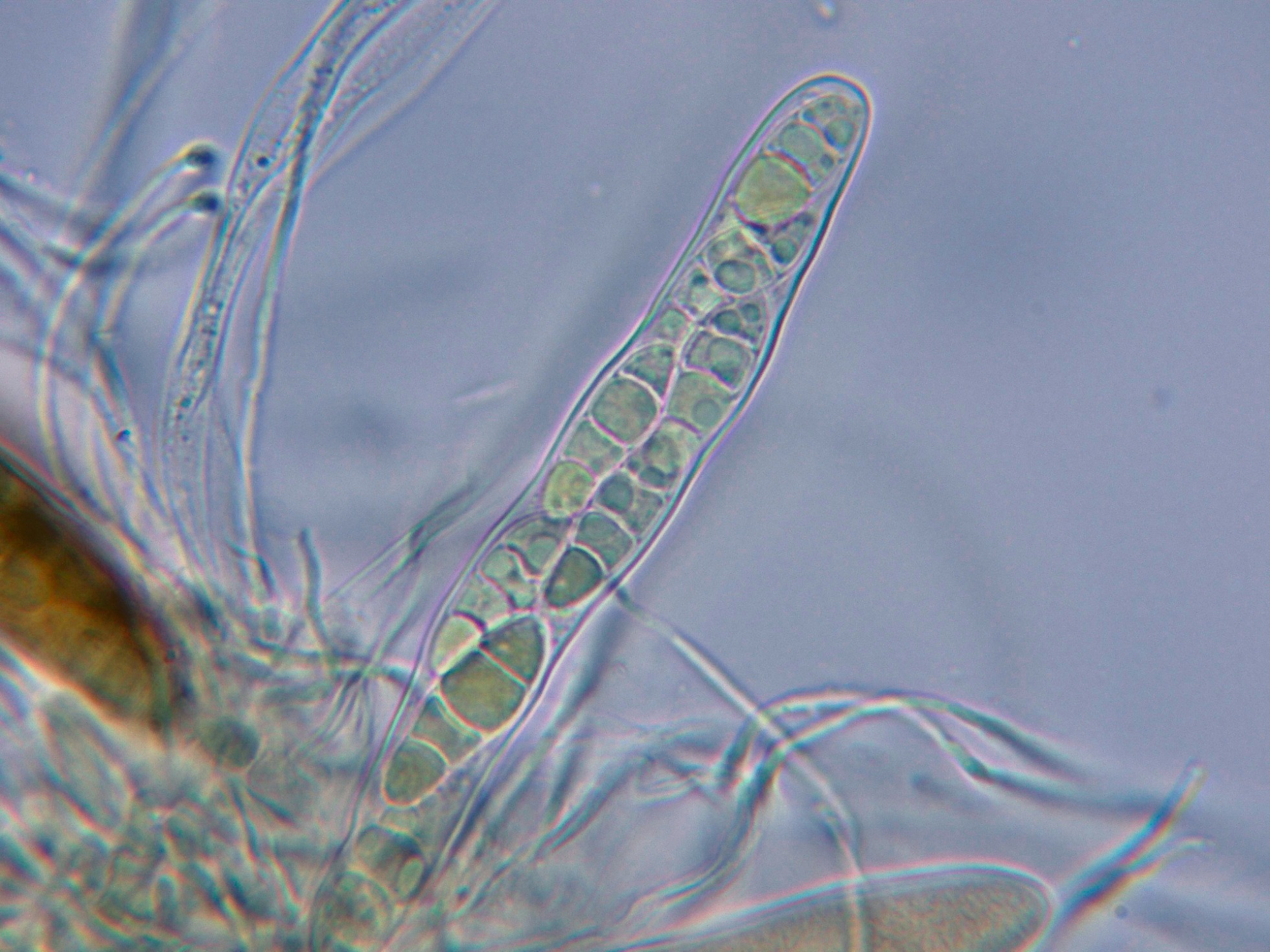
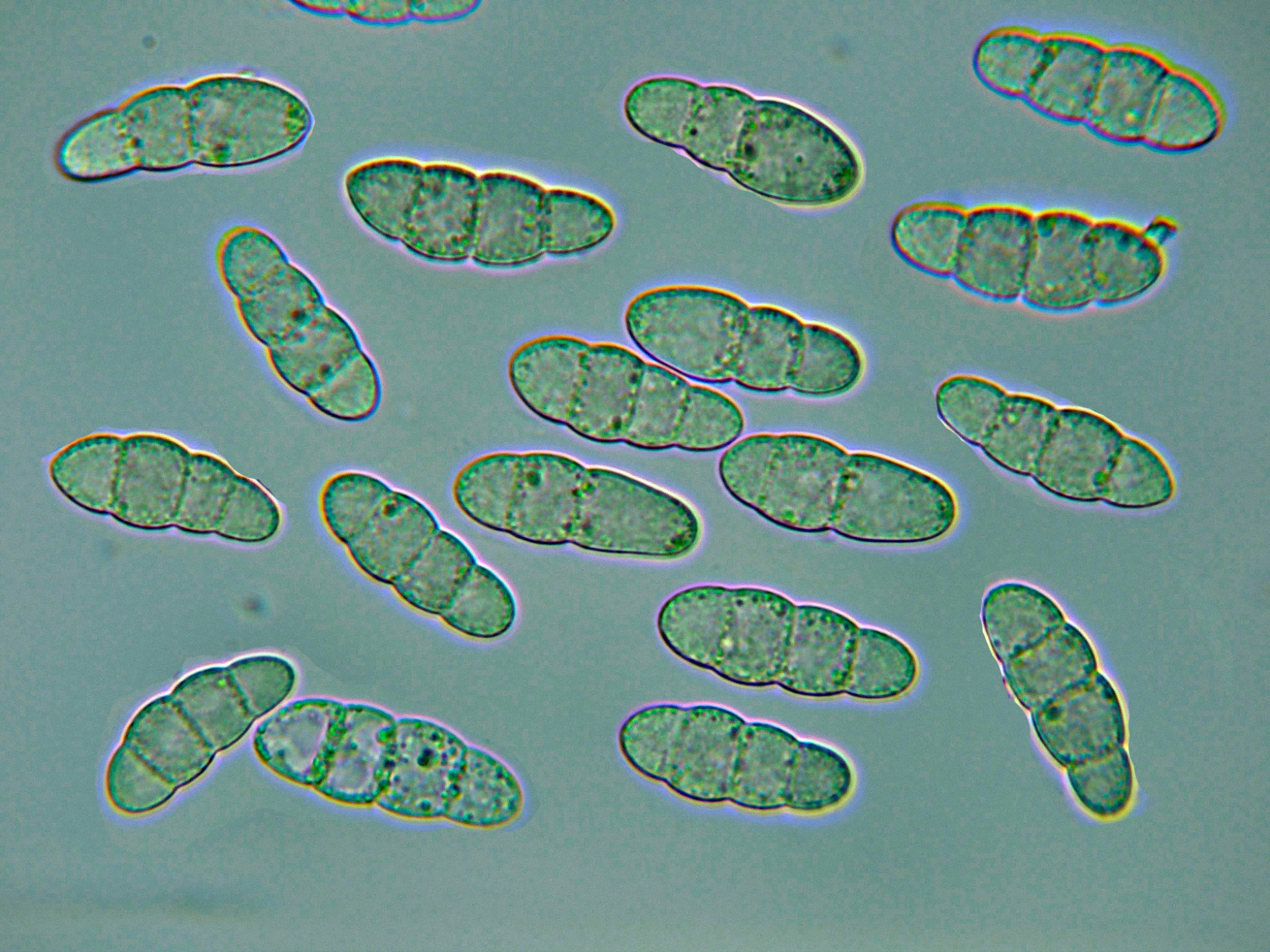
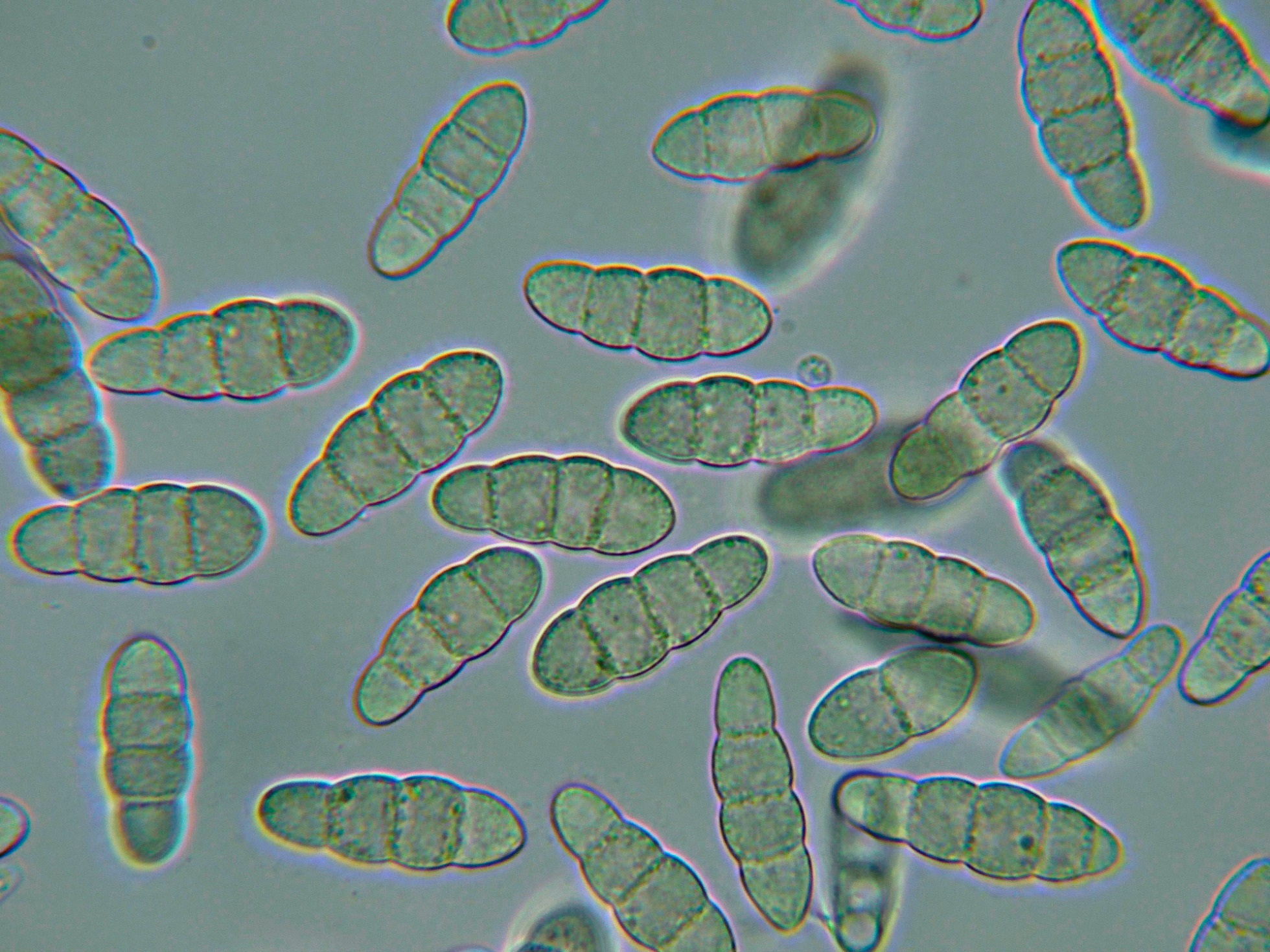
Ascosporas hialinas, mayoritariamente con tres septos, con un marcado constreñimiento en estos septos y unas medidas en agua de las esporas obtenidas por esporulación natural de:
(24.5) 25.3 - 29.2 (30.2) × (9) 9.3 - 10.9 (11.1) µm
Q = (2.5) 2.55 - 2.8 (2.9) ; N = 40
Me = 27.1 × 10.1 µm ; Qe = 2.7
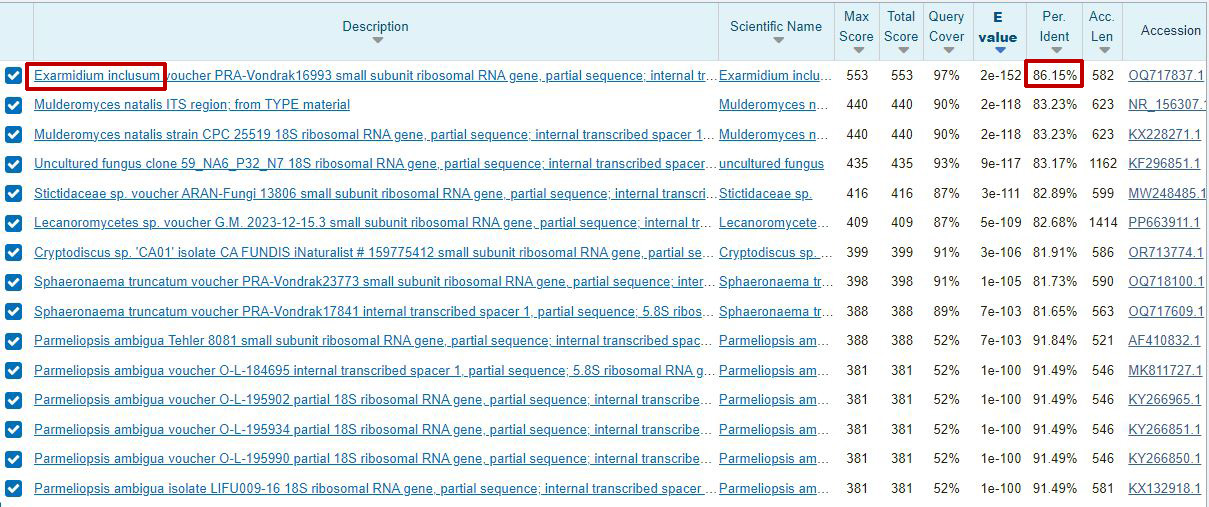
Como que con la microscopía no pude llegar a ninguna propuesta convincente, teniendo solo claro que estaba ante un Exarmidium, decidí probar con el análisis molecular, añado el resultado del Blast correspondiente al gen ITS:
Aquí vemos una coincidencia a nivel molecular por identidad del 86,15% con el Exarmidium inclusum, como que una coincidencia tan baja no aclara nada, es por lo que decidí probar con otros genes.
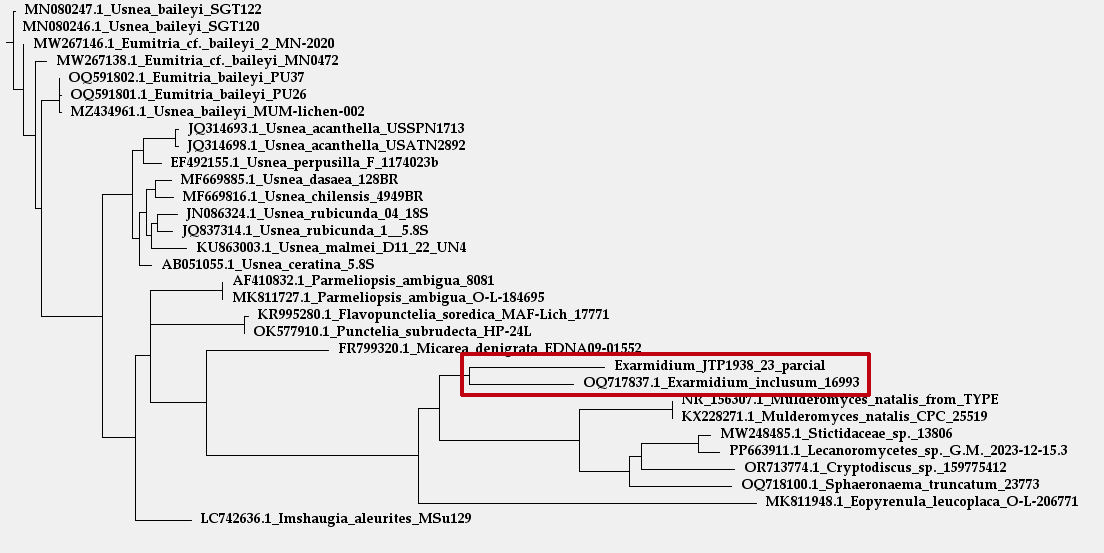
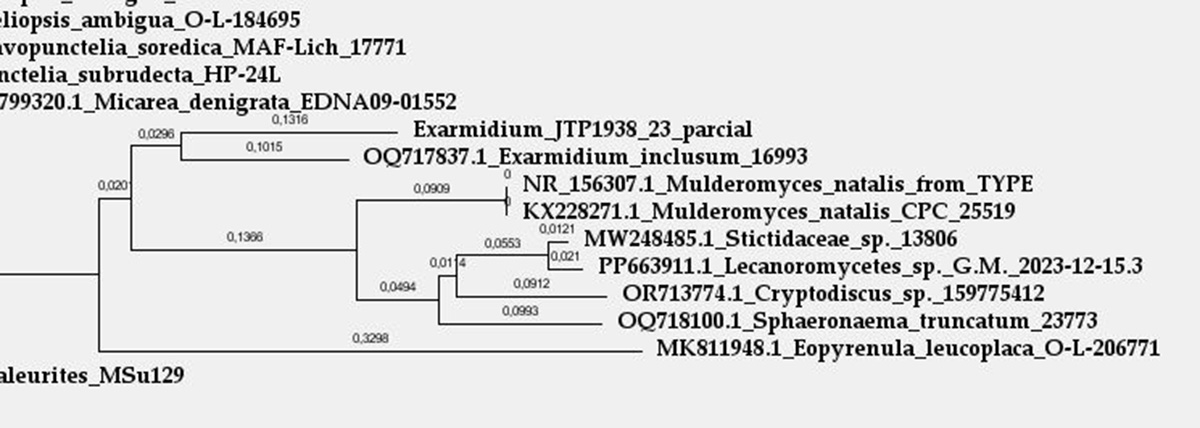
Añado una imágen del árbol filogenético del gen ITS:
Aunque coloca la especie de mi estudio en el mismo Clado que el Exarmidium inclusum, el árbol filogenético ya refleja una diferencia entre ambos.
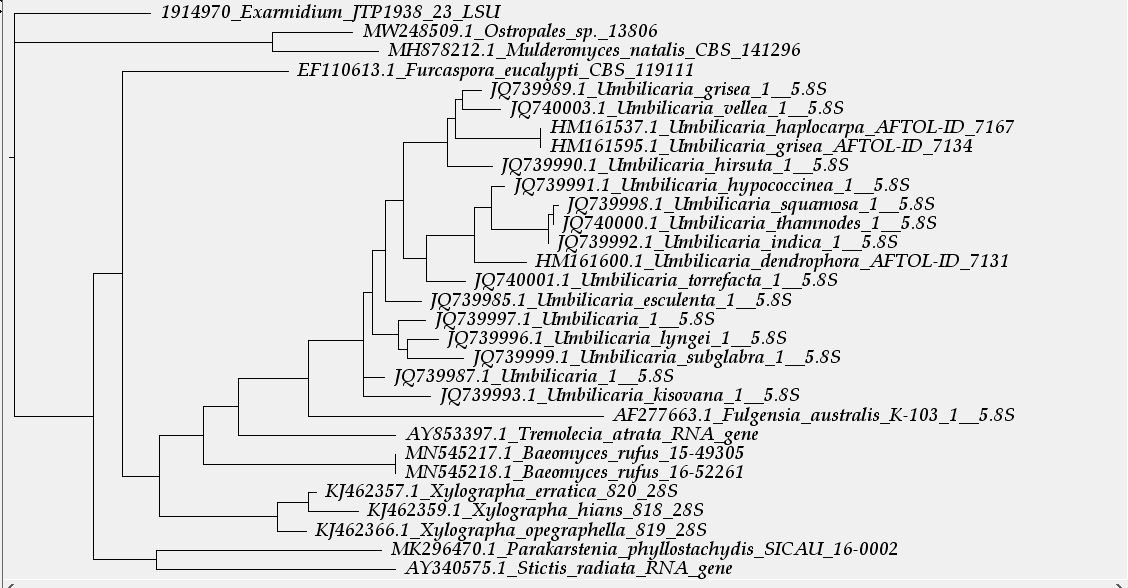
Añado el árbol filogenético del gen LSU:
Aquí el resultado refleja solo un acercamiento a nivel molecular con Ostropales spp., (parte superior de la imágen).
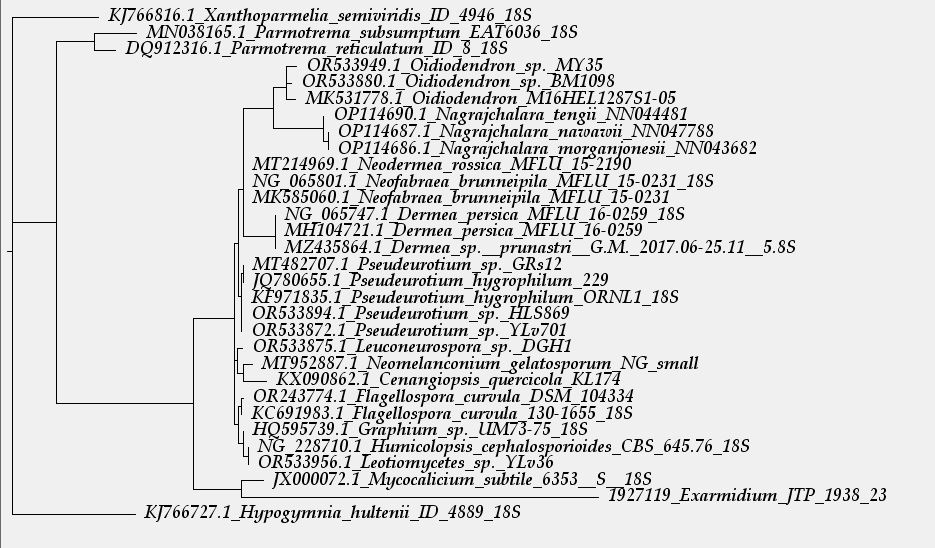
Ahora el árbol filogenético del gen SSU:
En este caso sitúa a mi especie en el mismo Clado que el Mycocalicium subtile (parte baja de la imágen), aunque muy alejado molecularmente de este.
Con todos estos datos obtenidos, tengo claro que a día de hoy solo disponemos de un registro en Genbank de un Exarmidium, y dentro del género solo hay uno del gen ITS del Exarmidium inclusum de una recolecta efectuada en la República Checa del 4 de abril de 2023, dejo una captura de pantalla del registro:
Llegado a este punto ya pruebo con los valores de la ramas con el sistema decimal:
Aquí ya se refleja una diferencia del Exarmidium de mi estudio con el Exarmidium inclusum superior al 0,03, algo que parece confirmar todas mis sospechas de que estamos ante una nueva especie aún no descrita.
Pidiendo consejo en privado a Baral, para poder comparar mi Exarmidium con el Exarmidium inclusum, consideró interesante el poder disponer de alguna otra secuencia del Exarmidium inclusum, para comparar con la recolecta de la República Checa, el Exarmidium inclusum creo haberlo estudiado el pasado año, dejo el enlace de mi trabajo:
www.fungipedia.org/setas-informacion-y-c...roscopia.html#108841
Como por supuesto seguía conservando el exsiccata, decidí mandar unas muestras a secuenciar, en un primer intento el resultado fue de "fallo" por ruido o contaminación, en un segundo intento la secuencia salió limpia, con el resultado de 100% Dacrymyces capitatus, evidentemente habían amplificado el ADN del Dacrymyces que brotaba justo al lado del Exarmidium inclusum, ahora ya me han confirmado del laboratorio de que ya no disponen de más material, con lo que les mandaré lo poco que me queda junto con mi próximo envío de muestras, así que ya seguiré informando en cuanto disponga de nuevos datos.
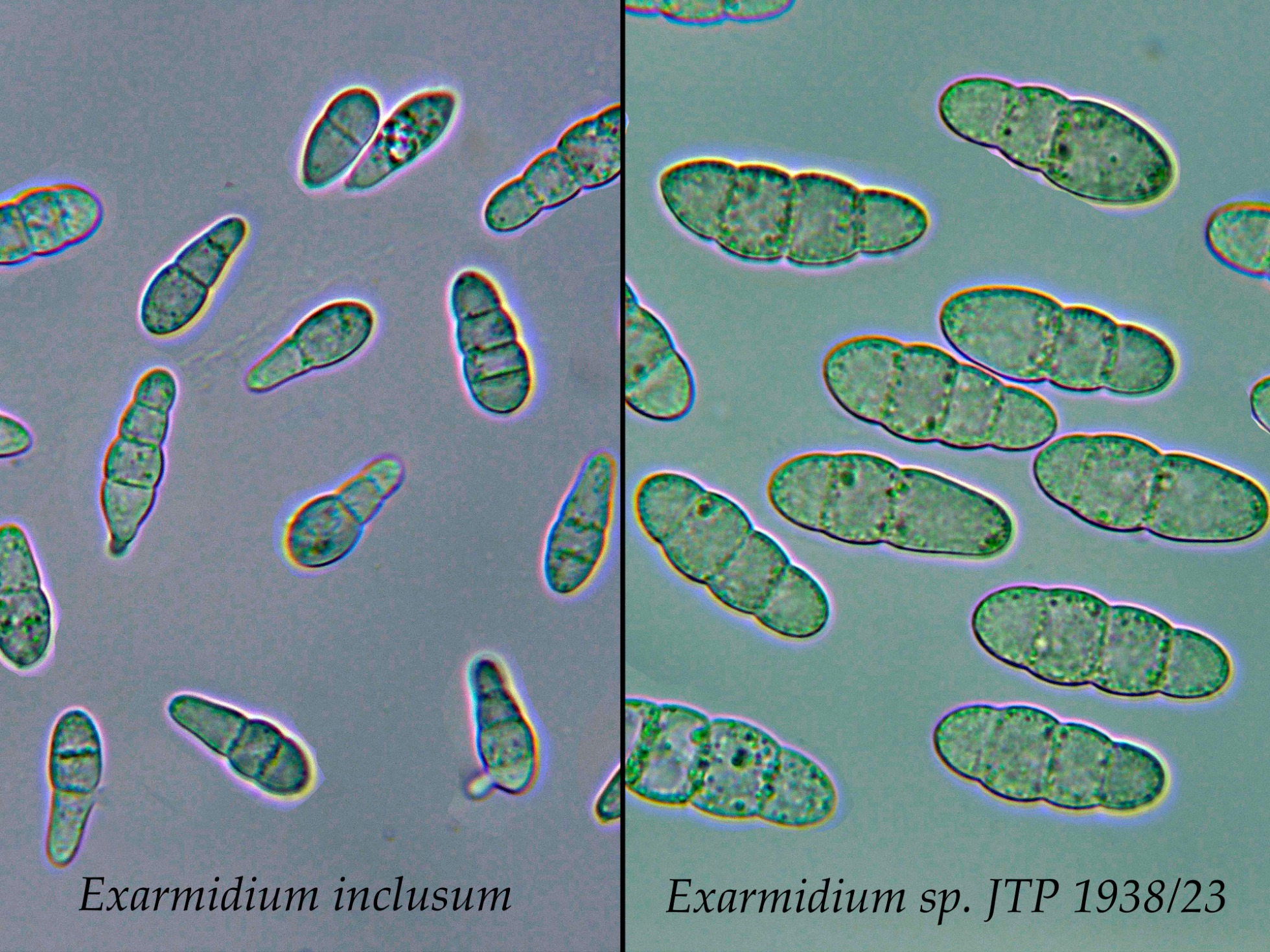
Para terminar añado una comparativa de los ascomas del Exarmidium inclusum y del Exarmidium del presente estudio a la misma escala, a mi propuesta la he llamado Exarmidium JTP 1938/23, que se corresponde con el número del herbario.
También una comparativa de las esporas de ambos a la misma escala:
Saludos a tod@s.
Un Exarmidium fotografiado el 1 de mayo de 2023, en un punto cercano a la localidad de Coll de Nargó (Lleida).
Unos pequeños apotecios brotando de manera dispersa pero abundante sobre una tabla de madera ya elaborada y trabajada y además bastante degradada de probablemente planifolio.
Los ascomas a 20 aumentos:
Otra imágen de los ascomas a 20 aumentos con la escala:
Los ascomas a 40 aumentos:
Estos ascomas a 40 aumentos con sus medidas:
La sección de los ascomas a 40 aumentos:
La sección de un ascoma con sus medidas:
Las hifas pigmentadas de la pared del peritecio:
El himenio a 400 aumentos en solución de Rojo Congo diluido:
Los ascos en solución de Rojo Congo a 1000 aumentos con unas medidas de (81.4) 82.6 - 108.1 (121) × (13.9) 14.7 - 17.3 (19) µm.
Los ascos en Reactivo de Melzer, con reacción débilmente dextrinoide:
Al utilizar Lugol, la reacción dextrinoide de los ascos se hace ya más evidente:
Los ascos en Lugol a 400 aumentos:
Los ascos en Lugol a 1000 aumentos:
Materia interascal a modo de paráfisis, muy densa, filiforme, con septos y una anchura de entre 1,85 a 2,45 µm.
La materia interascal con su anchura:
Ascosporas hialinas, mayoritariamente con tres septos, con un marcado constreñimiento en estos septos y unas medidas en agua de las esporas obtenidas por esporulación natural de:
(24.5) 25.3 - 29.2 (30.2) × (9) 9.3 - 10.9 (11.1) µm
Q = (2.5) 2.55 - 2.8 (2.9) ; N = 40
Me = 27.1 × 10.1 µm ; Qe = 2.7
Como que con la microscopía no pude llegar a ninguna propuesta convincente, teniendo solo claro que estaba ante un Exarmidium, decidí probar con el análisis molecular, añado el resultado del Blast correspondiente al gen ITS:
Aquí vemos una coincidencia a nivel molecular por identidad del 86,15% con el Exarmidium inclusum, como que una coincidencia tan baja no aclara nada, es por lo que decidí probar con otros genes.
Añado una imágen del árbol filogenético del gen ITS:
Aunque coloca la especie de mi estudio en el mismo Clado que el Exarmidium inclusum, el árbol filogenético ya refleja una diferencia entre ambos.
Añado el árbol filogenético del gen LSU:
Aquí el resultado refleja solo un acercamiento a nivel molecular con Ostropales spp., (parte superior de la imágen).
Ahora el árbol filogenético del gen SSU:
En este caso sitúa a mi especie en el mismo Clado que el Mycocalicium subtile (parte baja de la imágen), aunque muy alejado molecularmente de este.
Con todos estos datos obtenidos, tengo claro que a día de hoy solo disponemos de un registro en Genbank de un Exarmidium, y dentro del género solo hay uno del gen ITS del Exarmidium inclusum de una recolecta efectuada en la República Checa del 4 de abril de 2023, dejo una captura de pantalla del registro:
Llegado a este punto ya pruebo con los valores de la ramas con el sistema decimal:
Aquí ya se refleja una diferencia del Exarmidium de mi estudio con el Exarmidium inclusum superior al 0,03, algo que parece confirmar todas mis sospechas de que estamos ante una nueva especie aún no descrita.
Pidiendo consejo en privado a Baral, para poder comparar mi Exarmidium con el Exarmidium inclusum, consideró interesante el poder disponer de alguna otra secuencia del Exarmidium inclusum, para comparar con la recolecta de la República Checa, el Exarmidium inclusum creo haberlo estudiado el pasado año, dejo el enlace de mi trabajo:
www.fungipedia.org/setas-informacion-y-c...roscopia.html#108841
Como por supuesto seguía conservando el exsiccata, decidí mandar unas muestras a secuenciar, en un primer intento el resultado fue de "fallo" por ruido o contaminación, en un segundo intento la secuencia salió limpia, con el resultado de 100% Dacrymyces capitatus, evidentemente habían amplificado el ADN del Dacrymyces que brotaba justo al lado del Exarmidium inclusum, ahora ya me han confirmado del laboratorio de que ya no disponen de más material, con lo que les mandaré lo poco que me queda junto con mi próximo envío de muestras, así que ya seguiré informando en cuanto disponga de nuevos datos.
Para terminar añado una comparativa de los ascomas del Exarmidium inclusum y del Exarmidium del presente estudio a la misma escala, a mi propuesta la he llamado Exarmidium JTP 1938/23, que se corresponde con el número del herbario.
También una comparativa de las esporas de ambos a la misma escala:
Saludos a tod@s.
El siguiente usuario dijo gracias: Juan Andrés Román
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
- Juan Andrés Román
-

- Fuera de línea
- Spammer
-

Menos
Más
- Mensajes: 2555
- Gracias recibidas: 2753
8 meses 1 semana antes #110077
por Juan Andrés Román
Respuesta de Juan Andrés Román sobre el tema Una nueva especie de Exarmidium. Microscopía.
El siguiente usuario dijo gracias: JOSEP
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
- JOSEP
-
 Autor del tema
Autor del tema
- Fuera de línea
- Moderador
-

Menos
Más
- Mensajes: 8417
- Gracias recibidas: 8310
8 meses 1 semana antes #110083
por JOSEP
Respuesta de JOSEP sobre el tema Una nueva especie de Exarmidium. Microscopía.
Por favor, Identificarse o Crear cuenta para unirse a la conversación.
Tiempo de carga de la página: 0.059 segundos




























Foro de micología